Die Synthetische Biologie ist ein dynamisches Forschungsgebiet mit anwendungsbezogener Ausrichtung, zu dem Disziplinen mit biologischen, technischen und informationsverarbeitenden Schwerpunkten beitragen. Hauptforschungsfelder und Ziele der Synthetischen Biologie wurden bereits von der ZKBS vorgestellt (Stichwort Synthetische Biologie in der Rubrik Fokusthemen). Abbildung 1 zeigt wichtige Ereignisse in den Themenfeldern der Synthetischen Biologie zusammen mit Meilensteinen der Grundlagenforschung, die diese ermöglichten.
Synthetische Biologie
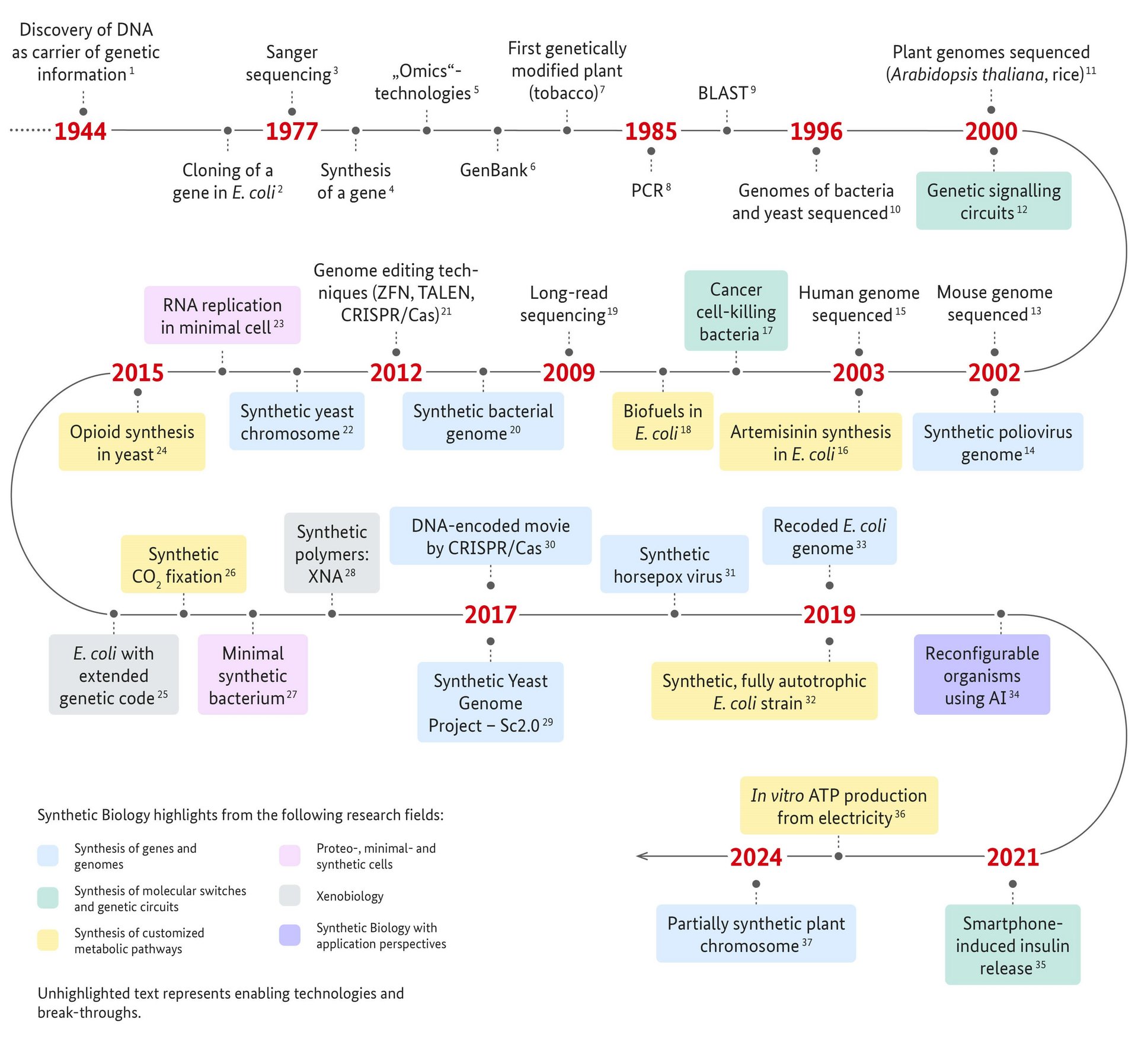
Abb. 1 Meilensteine der Synthetischen Biologie. Die Referenzen zu den einzelnen Highlights finden Sie unten auf der Seite. DNA - Deoxyribonucleic acid, E. coli – Escherichia coli, PCR - Polymerase chain reaction, BLAST - Basic local alignment search tool, TALEN - Transcription activator-like effector nuclease, CRISPR/Cas - Clustered Regularly Interspaced Short Palindromic Repeats/CRISPR-associated protein 9, RNA - Ribonucleic acid, CO2 - carbon dioxide, XNA - Xeno nucleic acid, AI - artificial intelligence, ATP - Adenosine triphosphate, ZFN – Zinc finger nucleases
Das Bundesministerium für Ernährung und Landwirtschaft (BMEL) hat die ZKBS mit einem Monitoring im Bereich der Synthetischen Biologie beauftragt, um die aktuellen Entwicklungen in den verschiedenen Forschungsbereichen sachverständig und mit Blick auf die biologische Sicherheit zu begleiten. Das Monitoring will rechtzeitig erkennbar machen, ob eine Anpassung der bestehenden Regulierungen erforderlich wäre.
Für die Sicherheitsbewertung der Synthetischen Biologie gibt es keine spezifische Regulierung in Deutschland oder Europa. Da die meisten Forschungsansätze in der Synthetischen Biologie gentechnisch veränderte Organismen (GVO) generieren, kann deren mögliches Risiko mit den bereits vorhandenen Methoden bewertet werden. Diese finden sich in den europäischen Richtlinien 2009/41/EC (Systemrichtlinie, contained use) und 2001/18/EC (Freisetzungsrichtlinie), die im deutschen Gentechnikgesetz (GenTG) umgesetzt worden sind. Die ZKBS prüft, ob die aktuellen Forschungsvorhaben vom Geltungsbereich des GenTG erfasst werden.
Ihren ersten Bericht zur Synthetischen Biologie in Deutschland [PDF, 101KB] veröffentlichte die ZKBS im Jahr 2012. Im Jahr 2018 erschien der zweite ZKBS-Bericht (nicht barrierefrei) [PDF, 1.21MB] (in Englisch mit deutscher Zusammenfassung), der den Stand der Forschung in den einzelnen Bereichen der Synthetischen Biologie weltweit zusammenfasst.
Die ZKBS führt seit Juni 2018 ein kontinuierliches Monitoring der Entwicklungen im Bereich der Synthetischen Biologie durch. Eine Auswahl relevanter Veröffentlichungen wird an dieser Stelle vorgestellt und regelmäßig aktualisiert. Basierend auf dem kontinuierlichem Monitoring wurde im Juli 2022 der dritte Bericht zur Synthetischen Biologie (nicht barrierefrei) [PDF, 839KB] veröffentlicht.
Alle drei ZKBS-Berichte (mit Stand Dezember 2021) sowie das kontinuierliche Monitoring (mit Stand Dezember 2023) stellen fest, dass die aktuellen internationalen Forschungsansätze der Synthetischen Biologie durch bestehende gesetzliche Vorgaben wie die europäischen Richtlinien und das GenTG reguliert werden und diesbezüglich aktuell kein Handlungsbedarf besteht.
Einzelne Teilbereiche der Erforschung von künstlichen Zellen, wie beispielsweise die Untersuchung von bakteriellen Zellteilungssystemen, finden in vitro, also außerhalb lebender Systeme, statt und werden daher nicht vom GenTG erfasst. Diese Versuche bergen bislang kein erkennbares spezifisches Gefährdungspotenzial; es handelt sich bei diesen Systemen nicht um lebensfähige Organismen.
Finden Sie mehr heraus
Quellenangaben zum Zeitstrahl der Synthetischen Biologie
1 Avery et al., 1944. Studies on the chemical nature of the substance inducing transformation of pneumococcal types. Induction of transformation by a desoxyriboenucleic acid fraction isolated from pneumococcus type III. J Exp Med 79 (2):137–58.
2 Chang & Cohen, 1974. Genome Construction Between Bacterial Species In Vitro: Replication and Expression of Staphylococcus Plasmid Genes in Escherichia coli. Proc Natl Acad Sci U S A 71(4):1030-4.
3 Sanger et al., 1977. DNA sequencing with chain-terminating inhibitors. Proc Natl Acad Sci U S A 74(12):5463-7.
4 Khorana et al., 1979. Total Synthesis of a Gene. Science 203(4381):614-25.
5 Review: Dai & Chen, 2022. Advances and Trends in Omics Technology Development. Front Med (Lausanne). 1(9): 911861.
6 https://www.ncbi.nlm.nih.gov/genbank/
Benson et al., 1993. GenBank. Nucleic Acids Res. 21(13):2963-5.
7 Bevan et al., 1983. A chimaeric antibiotic resistance gene as a selectable marker for plant cell transformation. Nature 304:184-7.
8 Patent US4683202A : Process for amplifying nucleic acid sequences. Filed Oct. 25, 1985; Date of Patent: Jul.28, 1987; Assignee: Cetus Corp, Inventor: Kary B. Mullis
Mullis et al., 1986. Specific enzymatic amplification of DNA in vitro: the polymerase chain reaction. Cold Spring Harb Symp Quant Biol. 51 Pt 1:263-73.
9 Altschul et al., 1990. Basic local alignment search tool. J Mol Biol 215(3):403-10.
10 Fleischmann et al., 1995. Whole-genome random sequencing and assembly of Haemophilus influenzae Rd. Science 269(5223):496-512.
Blattner et al., 1997. The complete genome sequence of Escherichia coli K-12. Science. 277(5331):1453-62.
Goffeau et al., 1996. Life with 6000 genes. Science 274(5287):546, 563-7.
11 The Arabidopsis Genome Initiative, 2000. Analysis of the genome sequence of the flowering plant Arabidopsis thaliana. Nature 408:796–815.
Eckardt , 2000. Sequencing the rice genome. Plant Cell 12(11):2011-7.
12 Elowitz & Leibler, 2000. A synthetic oscillatory network of transcriptional regulators. Nature 403(6767):335-8.
Review: MacDonald & Daens, 2016. Tools and applications in Synthetic Biology. Adv Drug Deliv Rev 105(Pt A):20-34.
13 Mouse Genome Sequencing Consortium, 2002. Initial sequencing and comparative analysis of the mouse genome. Nature 420:520–62.
14 Cello et al., 2002. Chemical synthesis of poliovirus cDNA: generation of infectious virus in the absence of natural template. Science 297(5583):1016-8.
15 International Human Genome Sequencing Consortium, 2004. Finishing the euchromatic sequence of the human genome. Nature 431(7011):931-45.
16 Martin et al., 2003. Engineering a mevalonate pathway in Escherichia coli for production of terpenoids. Nat Biotechnol. 21(7):796-802.
17 Anderson et al., 2006. Environmentally controlled invasion of cancer cells by engineered bacteria. J Mol Biol. 355(4):619–27.
Review: Chien et al., 2017. Advances in Bacteria Cancer Therapies using Synthetic Biology. Curr Opin Syst Biol 5:1-8.
18 Kalscheuer et al., 2006. Escherichia coli engineered for fuel production. Microbiology (Reading). 2006 Sep;152(Pt 9):2529-36.
Review: Clomburg at al., 2010. Biofuel production in Escherichia coli: the role of metabolic engineering and synthetic biology. Appl Microbiol Biotechnol 86(2):419–34.
19 Eid et al., 2009. Real-time DNA sequencing from single polymerase molecules. Science 323(5910):133-8.
Clarke et al., 2009. Continuous base identification for single-molecule nanopore DNA sequencing. Nat Nanotechnol. 4(4):265-70.
20 Gibson et al., 2010. Creation of a bacterial cell controlled by a chemically synthesized genome. Science 329(5987):52-6.
21 Kim et al., 1996. Hybrid restriction enzymes: zinc finger fusions to Fok I cleavage domain. Proc Natl Acad Sci USA 93(3):1156–60.
Christian et al., 2010. Targeting DNA double-strand breaks with TAL effector nucleases. Genetics. 186(2):757-61.
Jinek et al., 2012. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337(6096):816-21.
22 Annaluru et al., 2014. Total synthesis of a functional designer eukaryotic chromosome. Science 344(6179):55-8.
23 Caschera & Noireaux, 2014. Integration of biological parts toward the synthesis of a minimal cell. Curr Opin Chem Biol 22:85-91.
24 Galanie et al., 2015. Complete biosynthesis of opioids in yeast. Science 349(6252):1095-100.
25 Ostrov et al., 2016. Design, synthesis, and testing toward a 57-codon genome. Science 353(6301):819-22.
26 Schwander et al., 2016. A synthetic pathway for the fixation of carbon dioxide in vitro. Science 354(6314):900-4.
27 Hutchison et al., 2016. Design and synthesis of a minimal bacterial genome. Science 351(6280):aad6253.
28 Acevedo -Rocha & Budisa, 2016. Xenomicrobiology: a roadmap for genetic code engineering. Microb Biotechnol 9(5):666-76.
29 Richardson et al., 2017. Design of a synthetic yeast genome. Science 355(6329):1040-44.
30 Shipman et al., 2017. CRISPR-Cas encoding of a digital movie into the genomes of a population of living bacteria. Nature 547(7663):345-9.
31 Noyce et al., 2018. Construction of an infectious horsepox virus vaccine from chemically synthesized DNA fragments. PLoS ONE 13(1): e0188453.
32 Gleizer et al., 2019. Conversion of Escherichia coli to Generate All Biomass Carbon from CO2. Cell 179(6):1255-63.e12.
33 Fredens et al., 2019. Total synthesis of Escherichia coli with a recoded genome. Nature 569(7757):514.8.
34 Kriegman et al., 2020. A scalable pipeline for designing reconfigurable organisms. Proc Natl Acad Sci U S A 117(4):1853-9.
35 Mansouri et al., 2021. Smartphone-Flashlight-Mediated Remote Control of Rapid Insulin Secretion Restores Glucose Homeostasis in Experimental Type-1 Diabetes. Small 17(35): e2101939.
36 Luo et al.,2023. ATP production from electricity with a new-tonature electrobiological module. Joule 7(8): 1745-58.
37 Chen et al., 2024. A designer synthetic chromosome fragment functions in moss. Nat Plants 10(2):228-39.