Aktuelle Entwicklungen
- Was gibt es Neues aus der Forschung zur Synthetischen Biologie?
- Synthese von Genen und Genomen
- Synthese molekularer Schalter und genetischer Schaltkreise
- Synthese maßgeschneiderter Stoffwechselwege
- Proteo-, Minimal und synthetische Zellen
- Xenobiologie
- Methoden mit Einfluss auf die Synthetische Biologie
- Synthetische Biologie mit Anwendungsperspektiven
Was gibt es Neues aus der Forschung zur Synthetischen Biologie?
Die jährliche Zahl der Veröffentlichungen mit neuen Forschungsergebnissen hat inzwischen einen dauerhaft hohen Wert erreicht. Die ZKBS führt eine kontinuierliche Literaturrecherche zum Stichwort "Synthetische Biologie" durch. Dazu wird die Datenbank Pubmed (https://www.ncbi.nlm.nih.gov/pubmed) nach "synthetic biology" durchsucht und es werden die Newsletter relevanter Zeitschriften wie beispielsweise Nature, Science, ACS Synthetic Biology und andere thematisch orientierte Newsletter (z. B. The Scientist, SynBioBeta) gesichtet.
Die ZKBS wählt aus den Veröffentlichungen regelmäßig einige aus, die nach ihrer Ansicht für die einzelnen Forschungsfelder (Abb. 1) besonders typisch und relevant sind und stellt diese auf ihrer Homepage vor.
Mit Stand Dezember 2022 werden alle derzeitigen Forschungsansätze der Synthetischen Biologie durch bestehende gesetzliche Regelungen, insbesondere durch das GenTG, reguliert.
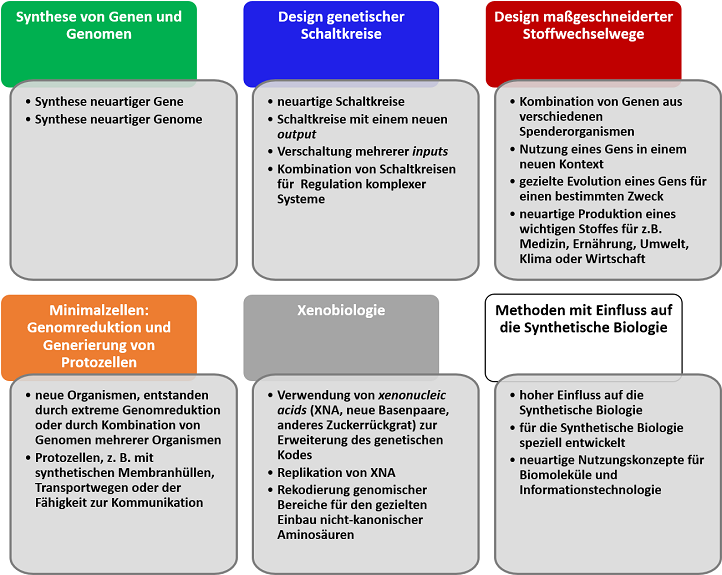 Die zentralen Forschungsfelder der Synthetischen Biologie (farbig unterlegt) und das neu hinzugekommene Feld „Methoden“ (weiß unterlegt). Zu jedem der Forschungsfelder werden exemplarische Forschungsschwerpunkte aufgelistet, zu denen wissenschaftliche Publikationen vorgestellt werden.
Quelle: © BVL
Die zentralen Forschungsfelder der Synthetischen Biologie (farbig unterlegt) und das neu hinzugekommene Feld „Methoden“ (weiß unterlegt). Zu jedem der Forschungsfelder werden exemplarische Forschungsschwerpunkte aufgelistet, zu denen wissenschaftliche Publikationen vorgestellt werden.
Quelle: © BVL
Die von der ZKBS ausgewählten Publikationen werden im Folgenden als Kurzzusammenfassungen vorgestellt.
(Stand des Monitorings: Dezember 2022)
Synthese von Genen und Genomen
2022
In diesem Themenbereich wurde 2022 nur eine Publikation ausgewählt, in der eine simultane Rekodierung des TAG-Stoppkodons gezeigt wird. Mithilfe informatischer und molekularbiologischer Methoden konnten in einer humanen Zelllinie 33 TAG-Stoppkodons gleichzeitig in die Sequenz TAA umgeändert werden (Chen et al. 2022).
2021
Image cell lineage with a synthetic recording system (Chow et al. 2021): Die Autoren entwickelten eine Methode, um die Abstammung einer Zelle in situ mit Hilfe eines bildgebenden readout aufzuzeichnen. Für ihr "integrase-editable memory by engineered mutagenesis with optical in situ readout (intMEMOIR)"-System verwendeten sie ortsspezifische Serin-Integrasen wie Bxb1. Dazu wurden die Zellen mit einem Barcode versehen, der von attP- und attB-sites flankiert ist. Diese sites ermöglichen eine Bxb1-spezifische Rekombination, die entweder zu einer Deletion oder einer Inversion des Barcodes führt. Der Zustand der Zellen kann mittels Fluoreszenz-in situ-Hybridisierung (FISH) ausgelesen werden. In einem Organismus können zehn orthogonale attP- und attB-sites verwendet werden, was die Aufzeichnung von 310 Zuständen ermöglicht. Um das System in vivo zu testen, haben die Autoren die Drosophila-Linie memoiphila hergestellt, die 10 Barcodes trägt und deren neuronale Zellen durch Hitzeschocks Integrase-editiert werden können. Die Editierung wurde vier Stunden nach der Eiablage eingeleitet, so dass die mit bestimmten Arrays markierten Neuroblasten ihre Editierung an alle Nachkommenzellen im erwachsenen Gehirn weitergeben können. Die erwachsenen Fliegen wurden anschließend seziert und ihre Gehirne in mehreren Runden einer FISH-Analyse unterzogen, bei der die induzierten Veränderungen sowie die Expression von acht endogenen Genen, die verschiedene neuronale Zelltypen markieren, ausgelesen wurden. Durch die Kombination von Genexpression und Zellklonen, die durch Barcode-Editierung identifiziert wurden, konnten mehrere bekannte Zelltypen bestimmt werden. Die Möglichkeit, die Abstammung einer Zelle direkt im nativen Gewebe sichtbar zu machen, könnte Aufschluss über die Rolle verschiedener Faktoren bei der Differenzierung von Zellen geben.
2020
Belcher et al. (2020) haben Bibliotheken von synthetischen Promotoren und korrespondierenden Transkriptionsfaktoren (TFs) für Pflanzen erstellt. Mithilfe der TFs soll eine präzisere Expression von Transgenen möglich werden und Pflanzen so leichter verändert werden können. Die Autoren begannen mit einem pflanzlichen Minimalpromotor, dem sie verschiedene verknüpfte cis-Elemente aus dem gut charakterisierten Hefe-GAL4-Regulon hinzufügten. Diese cis-Elemente werden durch den GAL4 TF gebunden. Die Promotoren mit den cis-Elementen zeigten unterschiedliche Expressionsstärken, die zur Feinregulierung der Genexpression genutzt werden können. 25 solcher Promotoren wurden in vivo erfolgreich in Arabidopsis thaliana getestet. Die Anzahl von TF wurde dann auf TFs aus anderen Familien und deren jeweilige cis-Elemente erweitert. Schließlich wurden der Promotor-Bibliothek Transaktivatoren und Repressoren hinzugefügt, indem bekannte Transaktivierungsdomänen aus Zea mays oder dem Herpes-Simplex-Virus Typ 1 oder die Repressordomäne SRDX mit den TFs oder verkürzten Versionen davon, die nur aus der DNA-Bindungsdomäne bestanden, fusioniert wurden. Die hier entwickelten hybriden Promotoren binden unterschiedliche TFs, die wiederum an verschiedene Aktivator- oder Repressor-Domänen gekoppelt sind. Dadurch können in Pflanzenzellen „Rechenoperationen“ mit multiplen Logikgattern ausgeführt werden. - zur Originalliteratur
Kotopka et al. (2020) etablierten ein in silico-basiertes Verfahren, um artifizielle Promotoren für Saccharomyces cerevisiae zu generieren. Um die Datengrundlage für dieses Modell zu generieren, wurden konservierte Motive von bekannten Promotoren in Kombination mit randomisierten spacer-Sequenzen verwendet, um zwei plasmidbasierte Bibliotheken in S. cerevisiae zu erstellen. Diese Bibliotheken umfassen über 675.000 konstitutive und über 327.000 durch einen künstlichen Transkriptionsfaktor (ZEV) induzierbare Promotoren. Die Bibliotheken wurden durch fluoreszenzaktivierte Zellsortierung in Kombination mit Hochdurchsatz-DNA-Sequenzierung (FACS-seq) analysiert. Im nächsten Schritt wurde ein in silico-Modell erstellt, das die Promotoraktivität als Funktion der Sequenz vorhersagt. Durch die Implementierung eines convolutional neural network (CNN) als deep-learning-Technik konnte das Modell die sehr großen Datenmengen verarbeiten. Um die mit diesem Modell getroffenen Vorhersagen zu validieren, wurden in silico-Sätze künstlicher Promotoren mit drei verschiedenen Sequenzdesigns erzeugt: 1) Screening: Generierung randomisierter Sequenzen basierend auf den Original-Bibliotheken, 2) Evolution: Mutagenese spezifischer Sequenzen und 3) gradient ascent: wiederholte Modifizierung initial randomisierter Sequenzen. Des Weiteren wurde bei der Promotoraktivitätsvorhersage eine GC-Einschränkung implementiert. Diese Sätze künstlicher Promotoren wurden im Vergleich mit Kontrollpromotorsätzen anschließend mittels Transformation in Hefe und FACS-seq-Analysen getestet. Dabei zeigte sich, dass die Screening- und Evolutionsdesign-Promotoren eine ähnlich hohe Diversität wie die stärksten Promotoren aus dem initial generierten Datensatz hervorbrachten. BeiF Promotoren, welche mit dem gradient ascent-Ansatz generiert wurden, war dies nicht der Fall. Diese verloren an Diversität. Weitergehende Optimierungen beim Evolutions- bzw. gradient ascent-Design führten letztlich zu expressionsstarken Promotorsequenzen, selbst wenn eine GC-Einschränkung implementiert wurde. Dieses Modell kann nicht nur verwendet werden, um Sequenzen mit nützlichen und seltenen Eigenschaften zu erzeugen, sondern auch, um große und sequenzvielfaltige Sätze von Promotoren mit hoher Aktivität zu generieren. - zur Originalliteratur
Lin et al. (2020) etablierten ein Einzelstrang-Doppelstrang-(ss-ds-)DNA-Gerüst für eine DNA-basierte, dynamisch operierendende und wiederverwendbare Datenspeicherung (DORIS). Systeme, welche DNA-basierte Datenspeicher nutzen, sollten drei grundlegende Kriterien erfüllen: 1) Skalierbarkeit, 2) Kompatibilität mit effizienter und dichter Kodierung und 3) Wiederverwendbarkeit. Um diese Kriterien zu erfüllen, wurde Doppelstrang-(ds-)DNA mit einem Einzelstrang-Überhang (ss-dsDNA) per PCR hergestellt. Dem Einzelstrang („file address“, 20 nt) folgte ein dsDNA-Anteil mit dem T7-Promotor (23 nt) und den kodierten Daten (data payload, 117 nt). Dabei sieht das Design die gleiche „file address“ für alle DNA-Stränge vor, die für eine bestimmte Datei kodieren. Dies ermöglicht es, die file-relevanten Stränge mittels magnetgekoppelter komplementärer Oligonukleotide aus einem Pool gemischter DNA-Moleküle bei Raumtemperatur zu separieren. Anders als bei PCR-basierten Separationsmethoden wird der data payload-Abschnitt nicht aufgeschmolzen, wodurch das Binden der Oligonukleotide an eventuelle ähnliche Sequenzen im data payload verhindert wird. Dies ermöglicht einen höheren Spielraum in der Gestaltung des data payload und erhöht somit die Informationsdichte (Information pro nt) und die Kapazität (Maximalspeicherung des Systems). Um als Datenspeicher fungieren zu können, wurde DORIS so konzipiert, dass auch während der Speicherung Änderungen wie das Sperren und Entsperren von Daten, die Änderung der Dateinamen oder das Löschen von Daten möglich sind. Dies wurde beispielhaft für die Datensperrung demonstriert, indem bei hohen Temperaturen (98°C) ein 20 nt langes, zur "file address" komplementäres Oligonukleotid an die ss-dsDNA angelagert wurde und diese somit verschlüsselte. Um das System zu entsperren, wurde ein „Schlüssel“-Oligo verwendet, welches komplementär zum „Sperr“-Oligonukleotid ist. Die Änderung der Dateinamen erfolgte ebenfalls über ein Oligonukleotid, in dem die neue „file address“ und die alte „file address“ in einer zusammengesetzten Sequenz vorliegen, wodurch letztlich eine neue Einzelstrang-Überhangsequenz erstellt wird. Das Löschen von Daten aus dem Speicher erfolgte durch Oligonukleotide, welche die „file address“ blockieren. Die erhöhte Informationsdichte und Kapazität des Systems, kombiniert mit der Möglichkeit, während der Speicherung der Daten Modifikationen vorzunehmen, stellt einen ersten Schritt in Richtung DNA-basierter paralleler Verarbeitung von extrem hohen Informationsmengen (z. B. medizinische, genombasierte und Finanzdaten) dar. - zur Originalliteratur
2019
Fredens et al. (2019) synthetisierten das bisher größte artifizielle Genom, ein Escherichia coli-Genom mit einer Größe von 4 Mbp. Dazu wurde mithilfe der REXER (replicon excision for enhanced genome engineering through programmed recombination)-Technik das Gesamtgenom von E. coli MDS42 durch ein synthetisches Genom ersetzt. Für das synthetische Genom wurden in den proteinkodierenden Genen nur 61 der 64 möglichen DNA-Kodons verwendet. Zwei Kodons für Serin (TCG und TCA) und ein Stopp-Kodon (TAG) wurden durch synonyme Kodons ersetzt, sodass insgesamt 18.214 Kodons geändert wurden. - zur Originalliteratur
Wang et al. (2019) entwickelten und erprobten ein System zur präzisen Aufteilung des unmodifizierten Genoms von E. coli auf definierte, zirkularisierte artifizielle Chromosomen. Dabei erfolgte die Aufteilung des E. coli-Genoms in der Zelle mittels Cas9, dem lambda-Red-Rekombinationssystem und einem artifiziellen bakteriellen Chromosom (BAC), welches als Akzeptor für einen Teil des Genoms fungiert. So entstand ein reduziertes Genom-Chromosom und ein BAC-Chromosom mit dem Restgenom. Die Aufteilung des Genoms hatte nur einen geringen Einfluss auf das Wachstumsverhalten der Zellen und blieb generationsübergreifend stabil. Zusätzliche Modifikationen des Systems erlaubten es, gezielte Inversionen bzw. Translokationen im getrennten Genom vorzunehmen und definierte Abschnitte von Genomen unterschiedlicher E. coli-Stämme miteinander zu kombinieren. Im Ergebnis wurde ein System geschaffen, dass ein schnelles und präzises engineering von großen Genomen ermöglicht. - zur Originalliteratur
Synthese molekularer Schalter und genetischer Schaltkreise
2022
Für diesen Bereich wurden zehn Publikationen ausgewählt.
Mehrere Publikationen befassen sich mit synthetischen Schaltern, die orthogonal zu zellulären Schaltern funktionieren. Beispielsweise wurde in Pflanzen ein Photorezeptor etabliert, der durch blaugrünes Licht aktiviert wird und auch in Pro- und anderen Eukaryoten anwendbar ist (Piccinini et al. 2022). Ein UV-Licht sensitiver Schalter funktioniert, indem eine nicht-kanonische Aminosäure in ein Enzym eingebaut wird, wodurch dieses Enzym nicht mehr durch seinen natürlichen Inhibitor feedback-inhibiert werden kann. Durch Bestrahlung mit UV-Licht wird eine sperrige o-Nitrobenzyl-Gruppe entfernt, sodass dass der Inhibitor wieder an das Enzym binden kann (Bhagat et al. 2022). Für Pflanzen wurden genetische Schaltkreise entwickelt, die eine spezifische Genexpression in verschiedenen Geweben erlauben. Diese Schaltkreise wurden dazu eingesetzt, das laterale Wachstum der Seitenwurzeln von Arabidopsis thaliana zu modifizieren (Brophy et al. 2022). Für die Anwendung in Pflanzen wurden auch orthogonale synthetische Promotoren entwickelt, die unter Kontrolle des endogenen Signalmoleküls Ethylen stehen (Kar et al. 2022). In der Krebstherapie wurden genetische Schaltkreise in Bakterien erforscht, die nach einer Temperaturerhöhung über Ultraschall zu einer gezielten Ausschüttung von Zytokinen führen (Chen et al. 2022). miRNAs werden in künstlichen Logikschaltkreisen, die verschiedene Rechenoperationen in Zellen ausführen können, verwendet. Die miRNAs binden dazu an doppelsträngige DNA-Stränge, sodass diese geöffnet und für weitere Interaktionen frei werden (Chen & Chen 2022). Neuronale Netzwerke wurden ohne die Nutzung von physischen Experimenten in Bakterien dazu programmiert, Rechenoperationen auszuführen oder auf Umweltbedingungen zu reagieren. Schließlich wurden die so getesteten Netzwerke auch in Bakterien implementiert (Becerra et al. 2022). In Säugerzellen wurden Logikgatter gebaut, die auf dem Einbau von zwei verschiedenen nicht-kanonischen Aminosäuren beruhen. Nur bei Vorhandensein einer oder beider nicht-kanonischer Aminosäuren sind diese Schaltkreise aktiv (Mills et al. 2021). Sogenannte Baktoneuronen wurden in Escherichia coli mit jeweils drei inputs und drei outputs konstruiert und bilden zusammen fünf artifizielle Neurosynapsen. Sie werden für zelluläre Rechenoperationen genutzt (Srivastava & Bagh 2022). Mithilfe eines Schaltkreises, bestehend aus Transkriptionsfaktoren, wurde in Säugerzellen Multistabilität erreicht. Dabei kann eine Zelle durch äußere Reize in Kombination mit dem eingebauten Schaltkreis einen bestimmten Zustand einnehmen (Zhu et al. 2022).
2021
LED control of gene expression in a nanobiosystem composed of metallic nanoparticles and a genetically modified E. coli strain (Aratboni et al. 2021): Die Autoren haben eine photothermische Kontrolle der Genexpression mit Hilfe von nichttoxischen metallischen Nanopartikeln, die Licht in Wärme umwandeln, etabliert. Freie Leitungselektronen in Goldnanopartikeln (AuNPs) schwingen bei Beleuchtung, und ein Teil dieser Energie wird als Wärme abgegeben und erhöht die Temperatur des umgebenden Mediums. Die Autoren aktivierten AuNPs mit einer Leuchtdiode in Mikroorganismen, die das fluoreszierende Protein mCherry unter Kontrolle eines U6-RNA-Thermometers exprimieren. RNA-Thermometer kommen natürlicherweise in den 5'-untranslatierten Regionen von mRNAs vor und steuern die Expression nachgeschalteter Gene durch temperaturbedingte Veränderungen ihrer Konformation. Bei niedrigen Temperaturen maskiert das RNA-Thermometer die Ribosomen-Bindungsstelle, bei höheren Temperaturen schmilzt die RNA-Sekundärstruktur lokal und ermöglicht die Ribosomenbindung. Es wurde gezeigt, dass mCherry bei Raumtemperatur nur exprimiert wird, wenn die AuNPs beleuchtet werden. Diese Arbeit bietet eine weitere Möglichkeit zur Kontrolle der Genexpression in biologischen Systemen mithilfe von Beleuchtung.
De novo design of a reversible phosphorylation-dependent switch for membrane targeting (Harrington et al. 2021): Die Autoren entwickelten einen minimalen de novo peptidbasierten molekularen Schalter, der auf einer heterodimeren coiled-coil-Anordnung basiert und bei Phosphorylierung und Dephosphorylierung zwischen einer monomeren und einer dimeren Konformation wechselt. Bei Kopplung an ein membranbindendes Peptid kann der Schalter zwischen einem membrangebundenen und einem gelösten Zustand wechseln. Ein solcher Schalter kann für die Transkriptionsregulierung und den Aufbau orthogonaler, proteinbasierter Schaltkreise verwendet werden.
T cell circuits that sense antigen density with an ultrasensitive threshold (Hernandez-Lopez et al. 2021): Die Autoren konzipierten T-Zell-Schaltkreise, um "off-tumor"-Toxizitäten zu verhindern, die bei der Krebsbehandlung mit chimären Antigenrezeptor (CAR)-T-Zellen häufig auftreten. Die Autoren entwickelten CAR-T-Zellen, die Krebszellen anhand der Antigendichte zuverlässig von normalen Zellen unterscheiden und so eine erhöhte Abtötungsrate von Krebszellen ermöglichen. Durch die Kombination eines synNotch-Rezeptors mit einem CAR wurden ultrasensitive T-Zellen mit einem zweistufigen Erkennungsschaltkreis entwickelt, die den humanen epidermalen Wachstumsfaktor-Rezeptor 2 (HER2) erkennen. Der synNotch-Rezeptor erkennt das Antigen mit niedriger Affinität und wirkt so als Filter für Zellen mit hoher Antigen-Dichte. Wenn der synNotch-Rezeptor aktiviert wird, induziert er die Expression eines CAR mit hoher Affinität, der nach seiner Aktivierung die T-Zell-Proliferation und die T-Zell-vermittelte Abtötung von Tumorzellen auslöst. Die neu entwickelten T-Zellen wurden an menschlichen Leukämiezellen (K562), die unterschiedliche Mengen an HER2 exprimieren, auf ihre Fähigkeit getestet, zwischen Zellen mit >106,5 und 104,5 HER2-Molekülen pro Zelle zu unterscheiden. Diese HER2-Expressionsniveaus entsprechen denen von HER2-exprimierenden Krebszellen bzw. normalem HER2-exprimierendem Gewebe. Die T-Zellen waren in der Lage, die vorgesehenen Diskriminierungsparameter zu erfüllen. In einer gemischten Kultur mit HER2-Zellen hoher Dichte (107) und niedriger Dichte (104,8) eliminierten die T-Zellen selektiv die Zellen mit der hohen HER2-Dichte und ließen die Zellen mit der niedrigen Dichte unversehrt. Um zu untersuchen, wie sich diese T-Zellen in einer komplexeren Umgebung verhalten, wurde immungeschwächten Mäusen ein K562-Tumor mit hoher HER2-Dichte auf der einen Seite und ein K562-Tumor mit niedriger HER2-Dichte auf der gegenüberliegenden Seite subkutan in die Flanken injiziert. Nachdem die Tumore etabliert waren, wurden die T-Zellen verabreicht und zeigten eine ausgeprägte Fähigkeit zur Unterscheidung verschiedener HER2-Dichten: Während Tumore mit hoher Dichte aus den Mäusen eliminiert wurden, wuchsen Tumore mit niedriger Dichte weiter. Diese ultrasensitive Unterscheidung zwischen Zellen mit unterschiedlicher Antigen-Dichte stellt ein wichtiges Instrument für die Behandlung solider Tumore mit T-Zellen dar.
Spatiotemporally confined red light-controlled gene delivery at single-cell resolution using adeno-associated viral vectors (Hörner et al. 2021): Die Autoren entwickelten ein virales Vektorsystem basierend auf dem Adeno-assoziierten Virus (AAV) für den Transfer genetischer Informationen in Zellen, welches durch Beleuchtung mit rotem Licht gesteuert wird. AAV-Vektoren transduzieren sowohl sich teilende als auch sich nicht teilende Zellen und werden häufig für die Gentherapie eingesetzt. Die lichtbasierte Steuerung der Systeme kontrolliert die Transduktion auf der Ebene des Zelleintritts. Der AAV-Vektor ist so konstruiert, dass er seinen natürlichen Zelleintrittsrezeptor Heparinsulfat-Proteoglykan nicht erkennen kann, sondern den Phytochrom-Interaktionsfaktor 6 (PIF6) aus Arabidopsis thaliana exprimiert. Um die Interaktion mit der Zielzelle zu ermöglichen, wird ein Adapterprotein benötigt. Das Adapterprotein besteht aus dem Phytochrom B von A. thaliana und einem Ankyrin-Repeat-Protein, das spezifisch für ein Zelloberflächenprotein ist. Bei Beleuchtung mit rotem Licht interagiert der Phytochrom-B-Teil des Adapterproteins mit PIF6 auf dem viralen Vektor, um den Vektor zur Zielzelle zu rekrutieren und die Transduktion einzuleiten. Bei Beleuchtung mit langwelligem rotem Licht wird der virale Vektor von der Zelle getrennt. Durch Umschalten des Adapterproteins können verschiedene Zelltypen angesteuert werden, und das System könnte z. B. für die Gentherapie in vivo eingesetzt werden.
An engineered protein-phosphorylation toggle network with implications for endogenous network discovery (Mishra et al. 2021): Die Autoren entwickelten einen bistabilen Kippschalter, der auf reversiblen Protein-Protein-Phosphorylierungsinteraktionen basiert. Der Schalter enthält zwei Interaktionskreise, die sich gegenseitig unterdrücken, und zwei Eingänge, um zwischen den beiden Zuständen des Systems zu wechseln. Das Proteinnetzwerk wird in Saccharomyces cerevisiae aus elf Proteinen aufgebaut, die entweder aus dem endogenen MAKP-Signalweg stammen oder als exogene chimäre Proteine konstruiert werden, die endogene Proteindomänen mit Domänen aus Arabidopsis thaliana und Mus musculus kombinieren. Die Interaktion zwischen den Proteinen wird durch Phosphorylierung reguliert: ein Protein des einen Kreises interagiert nur dann mit dem Protein des nächsten Kreises, welchen es unterdrückt, wenn es phosphoryliert ist. Das resultierende Kippschalter-Netzwerk weist eine hohe Sensitivität auf, kann schnell auf Eingangssignale reagieren und zeigt über Zellteilungen hinweg eine langfristige Bistabilität. Das Netzwerk ist in der Lage, outputs wie Fluoreszenz oder die Aufhebung der Zellteilung zu kontrollieren. Darüber hinaus entwickelten die Autoren einen Rechenalgorithmus, der 109.401 potenzielle endogene bistabile Signalwege identifizierte. 186 dieser Pfade wurden getestet, und fünf zeigten eine bisher unbekannte Bistabilität. Der auf Proteinen basierende Kippschalter könnte als empfindlicher Umweltsensor oder als mikroelektronisches Gerät für den Einsatz im Darm verwendet werden, wo er medizinische Zustände wie innere Blutungen erkennen könnte.
Light-controllable RNA-protein devices for translational regulation of synthetic mRNAs in mammalian cells (Nakanishi et al. 2021): Die Autoren schufen ein System zur optogenetischen Kontrolle der mRNA-Expression modifizierter RNAs, in die modifizierte Nukleoside eingebaut sind. Die Autoren entwickelten zwei auf Licht reagierende Aktivierungssysteme auf der Grundlage des caliciviralen VPg-basierten Translationsaktivators (CaVT). Bei einem System handelt es sich um einen gespaltenen CaVT, bei dem die RNA-Bindungsdomäne und die Translationsaktivierungsdomäne nur interagieren können, wenn ein Ligand durch Licht freigesetzt wird. Das zweite System verwendet ein mit einer destabilisierenden Domäne fusioniertes CaVT, das schnell abgebaut wird, wenn der Ligand nicht verfügbar ist, und stabilisiert wird, wenn der Ligand durch Licht freigesetzt wird. Das gespaltene CaVT hat den Vorteil, dass es gegenüber kürzeren Expositionszeiten mit den freigesetzten Liganden robuster ist, während das mit einer destabilisierenden Domäne fusionierte CaVT nicht nur aktiviert, sondern auch unterdrückt werden kann. Die kontrollierbare Expression modifizierter RNAs kann für die Gentherapie nützlich sein, da diese RNAs eine höhere Translationseffizienz, geringere Immunogenität und Zytotoxizität und kein Risiko einer Insertionsmutagenese aufweisen.
Wearable materials with embedded synthetic biology sensors for biomolecule detection (Nguyen et al. 2021): Die Autoren haben zellfreie, gefriergetrocknete, gentechnisch veränderte Polynukleotid-Schaltkreise in Materialien wie Textilien oder Silikone eingebettet und so tragbare Sensoren für den Nachweis von kleinen Molekülen, Nukleinsäuren oder Toxinen geschaffen. Sie entwickelten zunächst einen Sensor mit dem lacZ-Operon als output, der zum Nachweis verschiedener inputs verwendet wurde: (1) zum Nachweis von Anhydrotetracyclin über einen Transkriptionsfaktor-gesteuerten Schaltkreis, (2) zum Nachweis von Ebola-Virus-RNA über einen toehold-switch und (3) zum Nachweis des kleinen Moleküls Theophyllin über einen riboswitch. Sensorschaltungen wurden auch in faseroptische Textilien eingebettet, um den Nachweis von viraler (HIV) oder bakterieller (Borrelia burgdorferi) RNA über einen fluoreszierenden oder lumineszierenden output zu ermöglichen. Für den direkten Nachweis von Nukleinsäuren wurde ein Sensor verwendet, der auf einem programmierbaren CRISPR-System basiert. Bei diesem Sensor erkennt eine Cas12a-Protein-gRNA-Kombination eine Ziel-dsDNA und spaltet anschließend eine gequenchte ssDNA-Fluorophorsonde, was zu einem fluoreszierenden output führt. Das CRISPR-System wurde erfolgreich für den Nachweis von Resistenzmarkern von Staphylococcus aureus eingesetzt. Die Autoren nutzten ihr System auch, um einen in eine Gesichtsmaske integrierten Sensor für den Nachweis von SARS-CoV-2-RNA zu konstruieren, der eine vergleichbare Empfindlichkeit mit dem standardisierten RT-PCR-Test im Labor aufwies.
2020
Chen et al. (2020) entwickelten Logikgatter aus de novo-konstruierten Proteinheterodimeren. Zur Erstellung dieser Logikgatter wurden zunächst wesentliche Eigenschaften der spezifischen Proteinbausteine (Blöcke) definiert: 1) es sollte eine große Anzahl von orthogonalen Paaren geben, um die Gatter-Komplexität nicht zu beschränken, 2) die Blöcke sollten modular und ähnlich aufgebaut sein, um die Gatterkonstruktion zu vereinfachen, 3) einzelne Blöcke sollten in der Lage sein, an verschiedene Partner mit unterschiedlichen und regulierbaren Affinitäten zu binden und 4) die Interaktion der Blöcke sollte kooperativ sein, um zu gewährleisten, dass die Gatter-Aktivierung nicht empfindlich auf stöchiometrische Veränderungen der inputs reagiert. Um diese vier Eigenschaften in ihr Design zu integrieren, nutzten Chen et al. existierende Datensätze orthogonal konstruierter Heterodimere (DHD), welche alle eine identische Struktur aus vier Helizes aufweisen. Die einzelnen Monomere wurden mittels flexibler Linker so modifiziert, dass die Interaktionsdomäne von der Fusionsdomäne verdeckt wird und freie Energie benötigt wird, um diese zu exponieren. Dieser Aufbau stellt sicher, dass ein Logikgatter nur aktiviert werden kann, wenn sich die Bindungsenergien kooperierender DHD-Paare aufsummieren. Die Autoren zeigten, dass die DHD-Logikgatter mittels aufgereinigter Schaltkreiselemente im zellfreien System und in Hefe unter variablen experimentellen Bedingungen funktional sind. Dabei wurde ein fluoreszierendes Protein, dessen Expression unter Kontrolle der Logikgatter stand, als Reporter genutzt. Darüber hinaus wurden Logikgatter auf DHD-Basis mit mehreren inputs getestet, um die Skalierbarkeit des Systems aufzuzeigen. In einem proof-of-concept verfolgten Chen et al. das Ziel, die Expression für das Immun-Checkpoint-Gen für das T cell immunoglobulin and mucin domain-containing protein 3 (TIM3) zu reprimieren. Hierfür wurde ein „ODER“- Gatter erstellt, bei dem eines der Monomere mit der transcription activator like effector (TALE)-DNA Bindedomäne und das andere mit der Krüppel-associated box (KRAB) Repressordomäne gekoppelt wurde. In der Tat erfolgte keine Expression von TIM3, wenn sich die Monomere in räumlicher Nähe zueinander befanden. Somit ist ein Protein-Logikgatter entstanden, dass aufgrund seiner Flexibilität, Zusammensetzung und Skalierbarkeit eine posttranslationale Kontrolle einer Vielzahl von biologischen Funktionen ermöglichen könnte. - zur Originalliteratur
Frei et al. (2020) generierten miRNA-basierte inkohärente feedforward (iFF)-Schaltkreise, um das Expressionsniveau eines Fremdgens trotz Änderungen des endogenen zellulären Milieus stabil zu halten. Dadurch trägt diese Arbeit zur Entwicklung von robusten synthetischen Schaltkreisen in Säugerzellen bei. Um die notwendigen Daten für das Design der iFF-Schaltkreise zu generieren, untersuchten Frei et al. zunächst, welche Belastung transient exprimierte synthetische Schaltkreise für Säugetierzellen darstellen. Es wurde gezeigt, dass die exogene Genexpression in HEK293T- und H1299-Zellen einen negativen Einfluss auf die Proteinexpression der Wirtszelle hat. Außerdem zeigten die Autoren, dass eine miRNA-vermittelte Herabregulierung die metabolische Belastung eines Schaltkreises vermindern kann. Basierend auf diesen Daten wurde ein Modell für synthetische Schaltkreise etabliert, in dem die Expression eines Fremdgens von endogenen zellulären Veränderungen entkoppelt ist. Das Modell war in der Lage, sich an bestehende Schaltkreistopologien wie open-loop und iFF-Schaltkreise anzupassen, um Pools von gemeinsam genutzten und begrenzten Ressourcen einzubeziehen. Als proof-of-concept wurde ein iFF-Schaltkreis mit einer miRNA-vermittelten Herabregulierung in silico und in vitro getestet, bei dem miRNA-target sites verwendet wurden, die komplementär zu einer endogenen oder synthetischen miRNA sind. Wie erwartet reagierte der iFF-Schalkreis weniger empfindlich auf Veränderungen der verfügbaren Ressourcen. - zur Originalliteratur
Krawczyk et al. (2020) haben einen genetischen Schaltkreis entwickelt, in dem die elektrische Stimulation humaner Zellen die Expression eines Transgens bzw. Sekretion von Protein-Therapeutika aus intrazellulären Vesikeln auslöst. Dabei wird die elektronische Information über eine Depolarisierung der Zellmembran in Proteinproduktion und –sekretion umgewandelt. Die Autoren fügten den spannungsabhängigen L-Typ-Kalziumkanal in die Membran von Säugerzellen ein. Die Kalziumkanäle öffnen sich, wenn die Membran durch einen elektrischen Impuls depolarisiert wird und lassen Kalzium einströmen, wodurch die Expression eines Zielgens über den nuclear factor of activated T cells (NFAT) induziert wird. Als proof-of-concept wurden humane β-Zellen, die nach Kalziumeinfluss Insulin sekretieren, durch Expression des Ionenkanals elektrosensitiv gemacht. Die Zellen wurden dann in ein bioelektronisches Implantat integriert, welches kabellos stimuliert werden kann. Nach Implantierung in Mäuse mit Diabetes Typ I waren die elektrosensitiven Zellen in der Lage, auf einen elektrischen Impuls hin Insulin zu sezernieren und den Blutglukosespiegel auf einen normalen Wert abzusenken. - zur Originalliteratur
Lajoie et al. (2020) entwarfen co-lokalisationsabhängige Proteinschalter (Co-LOCKR), die Boolesche UND-, ODER- und NICHT-Verknüpfungen durchführen, um ein spezifisches Zell-targeting zu ermöglichen. So kann beispielsweise eine spezifische Kombination von Antigenen auf der Oberfläche einer Tumorzelle erkannt werden und eine anti-Tumor-Antwort eingeleitet werden. Die Arbeit basiert auf dem LOCKR-Schalter (Langan et al. 2019), bei dem ein Proteinkäfig durch ein verriegelndes Protein inaktiv gehalten wird, bis ein Schlüsselprotein bindet und die Interaktion mit dem Effektorprotein ermöglicht. Für Co-LOCKR wurde eine neue Version mit kürzeren Helices, einer verbesserten hydrophoben Packung und einem zusätzlichen Wasserstoffbrückenbindungsnetzwerk generiert. Um den Co-LOCKR-Käfig und die Schlüsselproteine an einer Zelle zu rekrutieren, die bestimmte Zielantigene exprimiert, wurden target-Domänen hinzugefügt. Die Autoren zeigten, dass der Proteinkäfig und der Schlüssel durch die target-Domänen in einem UND-Schaltkreis zu Her2- und EGFR-exprimierenden Zellen rekrutiert werden. Beim Hinzufügen eines zweiten Schlüssels wurde ein ODER-Schaltkreis eingeführt. Eine NICHT-Logik wurde durch Hinzufügen eines Köder-Proteins etabliert, das als molekularer Schwamm für den Schlüssel fungiert und mit einer target-Domäne gegen einen zu vermeidenden Oberflächenmarker fusioniert ist. In einem proof-of-principle-Experiment generierten die Autoren chimäre Antigenrezeptor (CAR)-T-Zellen, die ein exogen exprimiertes Protein auf Zellen erkennen. Die CAR-T-Zellen waren spezifisch gegen Tumorzellen gerichtet, die Her2 und EGFR exprimieren, wenn der Co-LOCKR, der Schlüssel, und die target-Domäne hinzugefügt wurden. In Experimenten, in denen CAR-T-Zellen mit UND/ODER- oder UND/NICHT-Co-LOCKRs und Tumorzellen, die das designierte Antigen aufweisen, co-kultiviert wurden, führte der Co-LOCKR den erwarteten Schaltkreis aus, was zur T-Zell-Proliferation bei einer spezifischen Antigenkombination führte. In einer Co-Kultur verschiedener Tumorzellen war Co-LOCKR in der Lage, eine Zelllinie anhand ihrer Antigenkombination zu identifizieren. - zur Originalliteratur
Pinto et al. (2020) charakterisierten eine Bibliothek mit 34 getrennten Inteinen (internal proteins). Inteine sind auto-katalytische Proteinsegmente, die sich selbst aus einem größeren Vorläuferpeptid ausschneiden. In diesem als Protein-Spleißen bekannten Prozess werden anschließend die angrenzenden Aminosäurereste durch neue Peptidverbindungen verknüpft und das Intein entfernt. Getrennte Inteine verbinden zwei Hälften eines Proteins, die von verschiedenen Genen exprimiert werden. Die Autoren nutzten ein getrenntes mCherry-Protein, um die Fähigkeit der Inteine zum cis-Spleißen (beide Proteinhälften auf dem gleichen Plasmid) und zum trans-Spleißen (Proteinhälften und getrennte Inteine werden auf unterschiedlichen Plasmiden exprimiert) unter gleichen Bedingungen untersuchen zu können. Sie identifizierten 15 zueinander orthogonale getrennte Intein-Paare, die sowohl cis- als auch trans-spleißen konnten. Zehn dieser Paare konnten in einem zellfreien System auch simultan genutzt werden. Die getrennten Inteine wurden anschließend in einem biologischen Schaltkreis genutzt, indem sie an getrennte transkriptionelle Regulatoren fusioniert und in Schaltkreisen mit zwei inputs und zwei outputs sowie solchen mit jeweils drei inputs und outputs genutzt wurden. In einer weiteren Anwendung wurden die getrennten Inteine genutzt, um große repetitive Proteine aus mehreren Peptiden zusammenzusetzen. So können die getrennten Inteine auch zur Herstellung von Biomaterialien genutzt werden. - zur Originalliteratur
Wiechert et al. (2020) haben induzierbare Promotoren für eine präzise induzierbare Expression entwickelt und getestet, die auf dem bakteriellen Mechanismus des xenogenen counter-silencing beruhen. Xenogene silencer sind Nukleoid-assoziierte bakterielle Proteine, die bevorzugt an horizontal erworbene AT-reiche DNA binden. Die Autoren verwendeten den xenogenen silencer CspS, ein von Prophagen kodiertes Protein aus Corynebacterium glutamicum. CspS agiert als silencer, indem es an einen Zielpromotor bindet, oligomerisiert und einen Nukleoproteinkomplex bildet, der die Transkription inhibiert. Um diese Hemmung zu regulieren, wurde ein synthetischer counter-silencer in die Promotorsequenzen eingebaut, und zwar die Operatorssequenz des Transkriptionsfaktor GntR. GntR bindet an den synthetischen counter-silencer-Promotor und interferiert mit dem Nukleoproteinkomplex, so dass die Transkription beginnen kann. Es wurden 44 synthetische counter-silencer-Promotoren identifiziert. Darüber hinaus wurde das System verwendet, um einen Kippschalter in C. glutamicum zu generieren, der zwischen Zellwachstum und L-Valin-Produktion umschaltet. - zur Originalliteratur
Williams et al. (2020) haben an der kombinatorischen Erkennung von Antigen-Mustern auf Tumorzellen gearbeitet, um Krebszellen spezifisch erkennen zu können. Die Autoren verwendeten mehrere synthetische Notch-Rezeptoren (synNotch), um eine Reihe von Rezeptoren und outputs flexibel zu Schaltkreisen zu verknüpfen. In einem drei-input-UND-Logikgatter erkennt der erste synNotch-Rezeptor das Antigen 1, wird aktiviert und induziert die Expression eines zweiten synNotch-Rezeptors. Wird dieser synNotch-Rezeptor durch das erkennende Antigen 2 aktiviert, wird ein chimärer Antigenrezeptor (CAR) exprimiert, der das dritte Antigen bindet und zur Abtötung der Zielzelle führt. Als Alternative zum drei-input-UND-Logikgatter testeten die Autoren auch ein drei-input-UND-NICHT-Logikgatter, bei dem ein synNotch zur Aktivierung eines CAR führt und ein dritter synNotch-Rezeptor, falls aktiviert, die Expression eines pro-apoptotischen Proteins induziert. In diesem Schaltkreis führt die Anwesenheit nur der ersten beiden Antigene zur Abtötung einer Tumorzelle, während die Anwesenheit von Antigen 3 die T-Zelle selbst zerstört. - zur Originalliteratur
2019
Aoki et al. (2019) - Basierend auf dem Ingenieursprinzip des integralen Reglers wurde erstmals ein geschlossener biologischer Regelkreis in lebende E. coli-Zellen integriert. Dazu werden die Bacillus subtilis-σ- und anti-σ-Faktoren (SigW und RsiW) genutzt. SigW aktiviert die Expression von araC sowie des gene of interest (GOI), das durch den Regelkreis stabil gehalten werden sollte. Der anti-σ-Faktor RsiW bindet und inaktiviert SigW, sodass weniger araC und GOI exprimiert werden. Da RsiW gleichzeitig unter Kontrolle eines araC-abhängigen Promotors steht, wird bei geringerer araC-Konzentration auch weniger RsiW gebildet. Hieraus ergab sich ein Regelkreis, der in Experimenten auch Störungen, wie Temperaturschwankungen, Stand hielt. Als proof-of-principle nutzen die Autoren das Methionin-Synthetasegen als GOI, ein Enzym, welches in Methionin-freiem Medium essentiell für das Wachstum der Zellen ist. Der Regelkreis führte zu einer stabilen Wachstumsrate der Zellen. - zur Originalliteratur
Chung et al. (2019) entwickelten einen genetischen Schaltkreis, der ein bestimmtes, auf der Oberfläche von Krebszellen überexprimiertes Protein (ErbB) erkennt und daraufhin die Apoptose der Krebszelle induziert. Das System besteht aus zwei Proteinen: einer Protease, die an eine Phosphotyrosin-Bindedomäne (PBD) fusioniert ist, und einem Protein, welches an die Zellinnenseite bindet und eine beliebige Fracht trägt. Die Protease bindet mithilfe der PBD nur an konstitutiv phosphoryliertes ErbB, wie es vor allem in Krebszellen vorliegt und kommt dadurch in räumliche Nähe zur Zellmembran. Die Protease kann dann die Fracht abspalten und beispielsweise die Apoptose der Zelle induzieren. Das System wurde bereits über einen Adeno-assoziierten Vektor in pankreatische Krebszellen und gesunde Hepatozyten eingebracht und eine selektive Apoptose der Krebszellen gezeigt. - zur Originalliteratur
Huang et al. (2019) - In der Therapie von soliden Tumoren werden vermehrt onkolytische Viren genutzt. Um eine höhere Tumorspezifität und eine verbesserte onkolytische Wirkung zu erreichen, erstellten Huang et al. programmierbare onkolytische Adenoviren. Diese enthalten einen Schaltkreis, bestehend aus einem tumorspezifischen Promotor, einer tumorspezifischen microRNA und einer hauptsächlich in gesunden Zellen exprimierten microRNA. Ist der tumorspezifische Promotor aktiviert, die tumorspezifische microRNA erhöht und die für gesunde Zellen spezifische microRNA wenig vorhanden, exprimiert das Adenovirus den Faktor E1 und einen Immuneffektor. Dadurch konnte sowohl eine verbesserte onkolytische Wirkung als auch eine hohe Gewebespezifität für hepatozelluläre Karzinome erreicht werden. - zur Originalliteratur
Langan et al. (2019), Ng et al. (2019) - Zur Implementierung einer synthetischen feedback-Kontrolle für endogene Signalwege und synthetische genetische Schaltkreise wurde im ersten Schritt ein de novo-Proteinsystem (LOCKR-Switch) entwickelt, welches zur Konformationsänderung fähig ist (Langan et al.). Das System besteht aus einem Bündel von sechs Helices, wovon fünf den Protein-Käfig bilden und die sechste Helix eine Protein-Falle mit integrierter Peptidsequenz zur Bindung des Zielmoleküls darstellt. In der Ursprungskonformation ist die sechste Helix an den Käfig gebunden und die Falle unzugänglich für das Zielmolekül. Die Zugabe eines zum Käfig homologen kompetitiven Schlüssels geht mit einer Änderung der Konformation der Helices einher, in welcher die Bindestelle für das Zielmolekül so exponiert wird, dass die Protein-Falle funktionell wird. Auf diesem System aufbauend implementierten Ng et al. eine synthetische feedback-Kontrolle für endogene Signalwege und synthetische genetische Schaltkreise. In der zweiteiligen Arbeit wurde zunächst ein degronLOCKR-Switch (Langan et al.) verwendet, um die native MAPK-Signalkaskade in Hefe so zu modulieren, dass ein erhöhter oder verringerter output der Signalkaskade induziert werden konnte. Der zweite Teil der Arbeit beschäftigte sich mit der Anwendung des degronSwitches in synthetischen genetischen Schaltkreisen. Hierfür wurde in Hefe eine einfache, hormonell beeinflussbare, synthetische Transkriptionskaskade mit und ohne feedback-Kontrolle getestet, die aus zwei induzierbaren degronSwitch-fusionierten Transkriptionsfaktoren (GEM, Z3PM9) besteht. Im direkten Vergleich mit dem Schaltkreis ohne feedback-Kontrolle war der Schaltkreis mit feedback-Kontrolle anpassungsfähiger gegenüber äußeren Einflüssen (Hormonkonzentrationsveränderungen) und stellt somit die Grundlage zur Implementierung komplexer synthetischer Funktionen in Zellen dar. - zur Originalliteratur: Langan et al. (2019), Ng et al. (2019).
Liao et al. (2019) - Komplexe Schaltkreise gehen in Zellen oft durch evolutionäre Prozesse verloren, da sie mit einem Fitnessverlust einhergehen. Um diese Verluste zu vermeiden, nutzen die Autoren eine Ko-Kultur mit drei nacheinander eingesetzten E. coli-Stämmen, die jeweils ein Toxin-Antitoxin-System (TA-System) enthalten. Neben dem jeweils individuellen TA-System kodiert ein solcher Stamm auch für ein Antitoxin eines der anderen Stämme. Dadurch wird sichergestellt, dass Bakterien ohne das passende Antitoxin in der Ko-Kultur schnell abgetötet werden und von solchen mit dem Antitoxin ersetzt werden. Somit ist der neu hinzugegebene Bakterienstamm mit dem Antitoxin resistent gegenüber den zuvor gewachsenen Bakterien und kann nun die Kultur dominieren. In einem Rotationsprinzip könnte der zweite Stamm dann durch einen dritten ersetzt werden und dieser wiederum durch den ersten, bevor Mutationen zu unerwünschten Effekten führen. Durch dieses Prinzip bleibt der zweite Schaltkreis, der in allen drei Stämmen auf dem gleichen Plasmid wie das TA-System enthalten ist, stabil in der Kultur erhalten. Als proof-of-principle wurde ein Schaltkreis zur populationsabhängigen Lyse verwendet, der bei ausreichend hoher Konzentration eines quorum sensing-Moleküls die Lyse der Zellen auslöst. Normalerweise herrscht bei diesem Schaltkreis ein hoher Selektionsdruck, sodass sich die Zellen nach zwei Tagen adaptieren und keine Lyse mehr stattfindet. Durch das Rotationsprinzip im TA-System konnte die Bildung von Mutanten verhindert und die Langlebigkeit des Schaltkreises deutlich verlängert werden. - zur Originalliteratur
Pandi et al. (2019). Zur Erstellung von komplexen Schaltkreisen für die Synthetische Biologie, entwickelten Pandi et al. Signalwandler-Aktuator-Modelle in silico und testeten diese in Escherichia coli. Dabei bestehen die Modelle aus einem Signalwandler, welcher für das Gen eines Enzyms kodiert, welches ein Eingangssubstrat (z. B. Hippursäure, Kokain) in einen Ausgangsmetaboliten (z. B. Benzoat) umwandelt. Der Ausgangsmetabolit fungiert wiederum als Aktuator und reguliert die Expression eines Ausgangssignales (GFP). Durch die Kombination von zwei oder mehr Enzymgenen zur Konvertierung von Substraten wie z. B. Hippursäure oder Benzaldehyd in einem Operon entsteht ein analoger metabolischer Konzentrationsaddierer. Durch diesen Konzentrationsaddierer verstärkt sich die Fluoreszenz, wenn die Konzentration bereits eines der Metaboliten steigt. Das Signalwandler-Aktuator-System wurde zusätzlich in einem zellfreien System untersucht, in welchem der Signalwandler durch Zugabe verschiedener Konzentrationen der Enzym-kodierenden DNA gewichtet wurden. Dabei korreliert die Menge an zugegebener Enzym-DNA direkt mit der GFP-Fluoreszenz: je mehr Enzym-DNA zugegeben wird, desto stärker ist das GFP-Fluoreszenzsignal. Dieses gewichtete Signalwandler-Aktuator-System wurde als ein einlagiges metabolisches Perzeptron definiert. Das Perzeptron ist ein biologischer Rechenalgorithmus, welcher die Fähigkeiten der Neuronen zur Informationsverarbeitung, zum Lernen und zur Entscheidungsfindung nachahmt. Ähnlich wie Neurone sollten metabolische Perzeptrone in der Lage sein, mehrere Eingangssignale in Form von Substraten zu empfangen und diese, in Abhängigkeit von der gewichteten Summe der Eingangssignale, in einen Signalausgang, und zwar in Form einer spezifischen Transkription eines Zielgens, umzuwandeln. Das Perzeptron ist per se ein binärer Klassifizierer. Dies bedeutet, dass die Entscheidung AN getroffen wird und es zur Expression des Fluorophors kommt, wenn die berechnete gewichtete Summe einen vordefinierten Schwellenwert erreicht. Andernfalls bleibt das System auf AUS. Die Autoren erstellten ein metabolisches binäres Logikgatter-Perzeptron mit vier Eingängen, welches durch die unterschiedlichen Gewichtungen der einzelnen Detektoren in der Lage ist, unterschiedliche Mengen von Eingangssubstraten zu klassifizieren. In vitro-Experimente bestätigten, das mit dem in silico-Modell genaue Vorhersagen über die entsprechenden Gewichtungen getroffen werden können, um das vollständige ODER Logikgatterverhalten zu erreichen. In dieser Arbeit wurden eine analoge Signalverarbeitung und ein digitales Ausgangssignal so miteinander kombiniert, dass zukünftig komplexe Schaltreise entwickelt werden können, die eine schnelle und skalierbare Multiplex-Signalerfassung ermöglichen. - zur Originalliteratur
Saltepe et al. (2019) - Nanopartikel (NP) spielen zunehmend eine größere Rolle in klinischen Anwendungen und können unter Anderem zum Transfer von Chemotherapeutika in Tumorgewebe verwendet werden. In der Entwicklung neuer NP sind vor allem die Zytotoxizität und die Biokompatibilität von essentieller Bedeutung. Um für ein erstes Testverfahren einen zuverlässigen und schnellen Toxizitätssensor für das NP compound screening zu generieren, etablierten Saltepe et al. in E. coli einen genetischen Schaltkreis auf Basis des heat shock response-Mechanismus (HSR) von Mycobacterium tuberculosis. Dieser Mechanismus reagiert auf den durch toxische NP induzierten zellulären Stress mit der Aktivierung des HSR-Promotors und der Expression eines nachgeschalteten fluoreszierenden Reportergens. Im Ergebnis ist ein Test entstanden, welcher ein schnelles und einfaches optisches Auslesen ermöglicht. - zur Originalliteratur
2018
Purcell et al. (2018) stellen zwei Ansätze vor, mit denen das Design synthetischer Schaltkreise verschlüsselt werden kann, um den Schutz intellektuellen Eigentums sicherzustellen. Es soll verhindert werden, dass der Aufbau der genetischen Schaltkreise durch die Sequenzierung des Gesamtgenoms der Organismen entschlüsselt wird. Ein Ansatz nutzt ortsspezifische, unidirektionale Rekombinasen und deren Erkennungssequenzen, um die Anordnung des Schaltkreises zu verändern, wenn dieser nicht in Benutzung ist. Der zweite Ansatz basiert darauf, dass zusätzliche, für den Schaltkreis nicht wichtige Gene, als eine Art Camouflage verwendet werden. Nach Zugabe eines „molekularen Schlüssels“ werden die Effekte dieser Gene entfernt und der korrekte Schaltkreis funktioniert. Dieser Schlüssel kann zum Beispiel ein Plasmid sein, welches die Komponenten für eine CRISPR-Interferenz enthält und so zu einer Repression (der Expression) der Camouflage Gene führt.- zur Originalliteratur
Segall-Shapiro et al. (2018) haben einen inkohärenten feedforward loop entwickelt, der eine konstante Genexpression durch einen bestimmten Promotor bewirkt, unabhängig von der Kopienzahl des Plasmides oder der Lokalisation im Genom. Dieses System soll genutzt werden, wenn ein Gleichgewicht in der Genexpression verschiedener Enzyme, z. B. für einen maßgeschneiderten Stoffwechselweg, wichtig ist. Da Zellen im Wachstum dynamischen Einflüssen unterliegen, müssen ansonsten nach dem Einbringen eines Stoffwechselweges beispielsweise von einem Plasmid in ein Genom oft aufwändige Anpassungen vorgenommen werden. Die Autoren haben einen Repressor genutzt, der an den Promotor des Zielgens bindet. Das Gen für diesen konstitutiv exprimierten Repressor wurde vor das Zielgen kloniert, sodass beide Gene denselben Einflüssen und Störungen unterworfen sind und die Expression unabhängig von der Kopienzahl reguliert wird. - zur Originalliteratur
Toda et al. (2018) beschreiben ein synthetisches Netzwerk der Zell-Zell-Kommunikation, welches maßgeschneiderte komplexe und auch asymmetrische multizelluläre Strukturen generiert. Damit sollen synthetische, selbst-organisierende Gewebe oder Biomaterialien hergestellt werden. Der Schaltkreis basiert auf der synNotch juxtacrine signaling-Plattform und wurde so konzipiert, dass spezifische Zell-Zell-Kontakte zu einer Änderung in der Cadherin-Zell-Adhäsion und dadurch zu einer Differenzierung sowie zur Produktion von neuen Zell-Zell-Signalen führen. - zur Originalliteratur
Synthese maßgeschneiderter Stoffwechselwege
2022
Für diesen Bereich wurden zwei Publikationen ausgewählt.
Die Plattform BIOPOLYMER umfasst gentechnisch veränderte Bakterienstämme, Vektoren, Promotoren sowie Biosynthesegene zur Synthese von Anthrazyklinen aus dem Actinomyceten Streptomyces coelicolor. Die Plattform soll zur Synthese von Designer-Anthrazyklinanalogen genutzt werden (Wang et al. 2022). Die Hefe Komagataella phaffii (Pichia pastoris) wurde so verändert, dass sie CO2 als einzige Kohlenstoffquelle nutzen kann und gleichzeitig organische Säuren wie Itaconsäure oder Milchsäure produziert (Baumschabl et al. 2022).
2021
Microbial synthesis of vanillin from waste poly(ethylene terephthalate) (Sadler & Wallace et al. 2021): Die Autoren entwickelten gentechnisch veränderte Escherichia coli, die Kunststoffabfälle in Vanillin umwandeln. Es konnte gezeigt werden, dass das aus Polyethylenglykol (PET) gewonnene Monomer Terephthalat (TA) über einen fünfstufigen enzymatischen Weg, der aus Enzymen von Bakterien (Comamonas sp., Nocardia iowensis und Bacillus subtilis) und einem Säugetier (Rattus norvegicus, Ratte) besteht, in Vanillin umgewandelt wird.
A new-to-nature carboxylation module to improve natural and synthetic CO2 fixation (Scheffen et al. 2021): Die Autoren haben einen neuartigen Reaktionsweg für die CO2-abhängige Assimilation der C2-Verbindung Glykolat in den zentralen Kohlenstoff-Stoffwechsel ohne Kohlenstoffverlust entwickelt. Die wenigen natürlichen Wege zur Umwandlung von C2 in C3-Metabolite, z. B. die Photorespiration, führen zu Kohlenstoffverlusten. Die Autoren haben den bisher hypothetischen Tartronyl-CoA (TaCo)-Weg entwickelt, einen synthetischen photorespiratorischen Bypass, der eine Alternative zur natürlichen Photorespiration darstellt. Bei diesem Weg wird Glykolat durch eine Drei-Enzym-Reaktion in Glykolyl-CoA, dann in Tartronyl-CoA und schließlich in Glyzerat umgewandelt. Eine gentechnisch veränderte Glykolyl-CoA-Synthetase aus Erythrobacter sp., NAP1, katalysiert die erste Reaktion, während eine Malonyl-CoA-Reduktase aus Chloroflexus aurantiacus den dritten Schritt katalysiert und Tartronyl-CoA in Glyzerat umwandelt. Das Schlüsselenzym des Reaktionsweges, eine Glycolyl-CoA-Carboxylase (GCC), war bislang noch nicht identifiziert. Die Autoren untersuchten Propionyl-CoA-Carboxylasen auf ihr Potenzial als GCC, da diese eine ähnliche Struktur aufweisen. Sie identifizierten die Propionyl-CoA-Carboxylase von Methylorubrum extorquens als Kandidat, führten Mutationen in das Gen ein und veränderten dieses mithilfe einer gerichteten Evolution so, dass ein Protein mit ähnlicher Effizienz wie die natürlich vorkommenden biotinabhängigen Acyl-CoA-Carboxylasen entstand.
Das synthetische GCC-Enzym erwies sich in in vitro-Experimenten in Kombination mit den beiden anderen Enzymen des TaCo-Wegs als funktionell und konnte mit anderen synthetischen CO2-Fixierungszyklen wie dem CETCH-Zyklus (Crotonyl-Coenzym A (CoA)/Ethylmalonyl-CoA/Hydroxybutyryl-CoA) kombiniert werden.
Optogenetic control of plant growth by a microbial rhodopsin (Zhou et al. 2021): Die Autoren haben ein optogenetisches Kontrollsystem in Pflanzen etabliert, welches deren Wachstum steuern kann. Mikrobielle Rhodopsine sind Proteine, die häufig als optogenetische Werkzeuge exprimiert werden, sich aber in Pflanzen nur schwer exprimieren lassen, da ihnen der essentielle Co-Faktor Retinal fehlt. Die Autoren verwendeten das grünlichtgesteuerte Anionenkanal-Rhodopsin ACR1 aus der einzelligen Alge Guillardia theta in Tabakpflanzen. Um das notwendige Retinal bereitzustellen, wurde die bakterielle ß-Carotin 15,15`-Dioxygenase MbDio als Fusionsprotein mit dem Chloroplasten-Transitpeptid RC2 ko-exprimiert, welches die Akkumulierung von Retinal im Chloroplasten ermöglicht. Die Expression dieser Proteine ermöglichte einen durch grünes Licht induzierten Anionen-Efflux durch die Zellmembran der Pflanzen und eine Depolarisierung des Membranpotentials.
In einem proof-of-principle-Experiment nutzten die Autoren das MbDio-RC2-ACR1-Konstrukt, um die Hypothese zu testen, dass das Wachstum der Pollenschläuche durch eine lokale Anionenkanalaktivierung und dem daraus resultierenden Spannungsgradienten gesteuert wird. Während eine vollständige Beleuchtung zu einem massiven Anionen-Efflux führte, der das Röhrenwachstum entweder nicht beeinflusste oder es stoppte, änderte eine lokale Beleuchtung die Wachstumsrichtung der Röhre weg von der Seite, auf der ACR1 aktiviert war, und bestätigte damit die Hypothese. Diese Arbeit bildet die Grundlage für ein leistungsfähiges Instrument zur Untersuchung der Signalübertragung in Pflanzen und könnte auch die Prüfung der chemisch-elektrischen Signalübertragung ermöglichen.
2020
Miller et al. (2020) entwickelten semi-synthetische Chloroplasten, für die photosynthetisch aktive Membranen in zellgroßen Mikrotropfen enkapsidiert wurden. Um ein Modul für die lichtabhängige Regeneration von Adenosintriphosphat (ATP) und Nicotinamidadenindinukleotidphosphat (NADP+) zu etablieren, wurden Thylakoidmembranen aus den Chloroplasten von Spinacia oleracea isoliert und auf ihre Fähigkeiten zur lichtabhängigen Reduktion von NADP+ zu NADPH und zur Regeneration von ATP überprüft. Da die Thylakoidmembranen, wie angenommen, NADPH und ATP produzierten, verwendeten Miller et al. diese Membranen nicht nur, um Energie für einzelne Enzymreaktionen zur CO2-Fixierung bereitzustellen, sondern auch für einen vollständigen synthetischen Stoffwechselzyklus zur kontinuierlichen Fixierung von CO2. Dieser synthetische Stoffwechselzyklus besteht aus drei Teilen: 1) dem artifiziellen, 16 Enzyme umfassenden Crotonyl-CoA/Ethylmalonyl-CoA/Hydroxybutyryl-CoA (CETCH)-Stoffwechselweg zur in vitro-Fixierung von CO2, 2) der Glyoxylat/Hydroxypyruvat-Reduktase aus Escherichia coli und 3) einer Crotonase aus Pseudomonas aeruginosa. Zur Herstellung der artifiziellen semi-synthetischen Chloroplasten wurde zusätzlich eine Mikrofluidik-Plattform entwickelt, mit deren Hilfe die metabolisch aktiven Mikrokompartimente automatisch in Wasser-in-Öl-Mikrotröpfchen zusammengesetzt werden können. Die Optimierung der Tröpfchenpräparation führte dabei zu einem komplexen System, das nicht nur wesentliche Eigenschaften der Photosynthese besitzt, sondern auch die Energieproduktion von Systemen mit nur einem Enzym übertrifft. Die hier vorgestellte Arbeit bildet eine wesentliche Grundlage für die Entwicklung eines vollständig und sich selbst erhaltenden synthetischen Organells. - zur Originalliteratur
Reifenrath et al. (2020) nutzen vom endoplasmatischen Retikulum abgeleitete Vesikel zur Kompartimentierung von Enzymen eines Stoffwechselweges in membranumschlossene Organelle. So können mögliche toxische Nebenwirkungen oder unerwünschte Reaktionen, die durch ein heterolog exprimiertes Protein auftreten, vermieden werden. Die drei Enzyme des für Hefe toxischen cis,cis-Mukonsäure (CCM)-Stoffwechselwegs wurden mit einem synthetischen Peptid fusioniert, das die selbstassemblierende Region des Mais-Speicherproteins gamma-Zein (Zera) enthält. Zera kann sowohl in Pflanzen als auch in heterologen Systemen sogenannte Proteinkörper induzieren, die aus dem endoplasmatischen Retikulum oder der Vakuole stammen. Die Zera-induzierten Vesikel mit heterologen CCM-Enzymen wurden in der Hefe Saccharomyces cerevisiae erzeugt und die Funktionalität des Stoffwechselweges in den Vesikeln gezeigt. - zur Originalliteratur
2019
Gleizer et al. (2019) - Die Autoren haben einen E. coli-Stamm hergestellt, der erstmals CO2 als einzige Quelle zur Produktion von Biomasse nutzt. Dadurch soll die Umwandlung von atmosphärischem CO2 in Nahrung, Treibstoffe oder Biochemikalien ermöglicht werden. Für die Kohlenstofffixierung nutzen die Bakterien den Calvin-Zyklus, wobei das elektrochemisch produzierte Molekül Formiat (HCOO-) der Energiegewinnung dient. Die Umwandlung von Formiat in ATP treibt den Calvin-Zyklus an, der CO2 in Zucker und andere organische Moleküle umwandelt. Die Autotrophie wurde durch eine Evolution unter Laborbedingungen erreicht: in einem Chemostat wurde die Xylosekonzentration reduziert, während ausreichend Formiat und CO2 vorhanden waren. Die Bakterien wurden durch verschiedene Mutationen autotroph, von denen viele in Genen mit metabolischer Verbindung zum Calvin-Zyklus lagen. - zur Originalliteratur
Luo et al. (2019) haben den kompletten Biosyntheseweg für einige komplexe Cannabinoide in der Hefe Saccharomyces cerevisiae generiert. Dazu wurde der Mevalonat-Stoffwechselweg der Hefe modifiziert und es wurden Gene aus der Cannabis-Pflanze sowie Gene aus verschiedenen Organismen in das Hefegenom eingebracht, die für einen Hexanoyl-CoA-Stoffwechselweg erforderlich sind. Die gentechnisch veränderte Hefe kann nun zur kontrollierten industriellen Herstellung von Cannabinoiden genutzt werden. - zur Originalliteratur
South et al. (2019) haben mithilfe eines synthetischen, effektiveren Wegs der Photorespiration das Wachstum und die Produktivität der C3-Pflanze Tabak auf dem Feld verbessert. Besonders bei hohen Temperaturen und Trockenheit stellen C3-Pflanzen (zu denen auch Weizen, Reis und Soja gehören) vermehrt die für Pflanzen toxische Verbindung Glykolat her, welches mittels Photorespiration zu nicht-toxischen Verbindungen abgebaut wird. Dadurch können die Erträge um bis zu 50 % sinken. Durch Einbringen eines synthetischen Glykolatstoffwechselwegs in Chloroplasten und die gleichzeitige Inhibition des natürlichen Photorespirationswegs, konnte die Produktion von Biomasse in homozygoten transgenen Linien um 40 % erhöht werden. - zur Originalliteratur
Proteo-, Minimal und synthetische Zellen
2022
Für diesen Bereich wurden neun Publikationen ausgewählt.
Synthetische Zellen bestehend aus Liposomen können in einem synthetischen Paarungssystem miteinander interagieren und auf diese Weise verschiedene genetische Schaltkreise aktivieren (Gaut et al. 2022). Ein Drehmotor bestehend aus DNA-Origami wurde in DNA kodiert und konnte sich in einem Reaktionsmix selbst assemblieren. Der Motor ist in seiner Leistung mit natürlichen Motoren wie der ATPase vergleichbar und kann sich in eine Richtung bewegen (Pumm et al. 2022). Große Lipidvesikel wurden als synthetische Zellen zur Produktion von pro-angiogenetischen Faktoren genutzt. In Mäusen konnten diese einen neovaskulatorischen Prozess in Gang setzen (Chen et al. 2022). Eine artifizielle pflanzliche Zellwand, bestehend aus Pektin und Hemizellulose, konnte auf einem synthetischen Polymer und auf einer Lipiddoppelmembran assembliert werden (Notova et al. 2022). Artifizielle Zellen wurden auch auf Basis von Komponenten lebender Zellen aufgebaut. Dazu wurden Koazervate aus einem Polymer und ATP gebildet und zwei Arten von Bakterien im Inneren der Vesikel und auf der Außenhülle gebunden. Nach dem Aufbrechen der Bakterien entstanden artifizielle Zellen mit einer äußeren Bakterien-abgeleiteten Hüllmembran und innenliegenden zellulären Subkompartimenten, in denen Transkription und Translation ausgeführt wurden (Xu et al. 2022). Eine künstliche Zellteilungsmaschinerie wurde mithilfe von stabilen artifiziellen Vesikeln, sogenannten Dendrimersomen, hergestellt. Die Dendrimersomen binden Proteine des MinCDE-Systems, welches wiederum FtsZ-Proteine zur Bildung eines kontraktilen Rings in die Mitte der Zelle dirigiert (Wagner et al. 2022). In einem anderen Ansatz wurde die komplette Zellteilungsmaschinerie von E. coli in Lipidvesikeln rekonstruiert. Dazu wurden ebenfalls Min- und FtsZ-Proteine eingebracht und eine anschließende Formveränderung der Vesikel konnte beobachtet werden (Kohyama et al. 2022). In einem Vesikel wurde ein zelluläres Skelett aus DNA aufgebaut. Die DNA-Filamente wurden in Form rigider Strukturen aufgebaut, sodass sie Ring-ähnliche Formen annahmen, und zudem an der Außenwand der Vesikel verankert und konnten die Vesikel von innen heraus deformieren (Jahnke et al. 2022). Das intrazelluläre Transportsystem bestehend aus einem kolloidalen Motor und selbstassemblierenden Lipidröhren wurde nachgebaut. Die Motoren bewegen sich lichtabhängig auf den Lipidröhren und können eine Fracht in Form eines Lipidvesikels in eine bestimmte Richtung transportieren (Ghellab et al. 2022).
2021
Light-powered reactivation of flagella and contraction of microtubule networks: toward building an artificial cell (Ahmad et al. 2021): Die Autoren versuchten, ein autarkes System mit einer ATP-gesteuerten Bewegung aufzubauen. Um dies zu erreichen, konstruierten die Autoren schaltbare photosynthetische Liposomen, die Bacteriorhodopsin und eine ATP-Synthase aus Escherichia coli enthalten, die bei Lichteinfall ADP in ATP umwandeln kann. Sie koppelten das Modul mit dem aus Chlamydomonas reinhardtii isolierten funktionellen Flagellenmodul und beobachteten die Umwandlung von Licht in mechanische Arbeit. In einem anderen Ansatz wurde das ATP-Regenerationsmodul mit Mikrotubuli und dem molekularen Motor Kinesin-1 verkapselt, und es wurden Kontraktionen des fadenförmigen Netzwerks bei Lichtstimulation beobachtet. Diese Arbeit ist ein weiterer Schritt auf dem Weg zu fortgeschrittenen synthetischen Zellen, die ein breites Spektrum an zytoskelettabhängigen motorischen Funktionen nutzen können.
Programmable microbial ink for 3D printing of living materials produced from genetically engineered protein nanofibers (Duraj-Thatte et al. 2021): Die Autoren entwickelten eine mikrobielle Tinte, die von gentechnisch veränderten E. coli produziert wird und funktionalisiert werden kann. Die Tinte besteht aus Nanofasern, die sich aus bakteriellen curli-Fasern zusammensetzen. Das curli-Faser-Gen CsgA wurde so verändert, dass die curli-Proteine an ihren Enden mit einer Buckel- und einer Loch-Protein-Domäne aus Fibrin fusioniert sind. Diese Domänen ermöglichen die Selbstorganisation zu Nanofasern, die in ein 3D-Hydrogel gedruckt werden können. Das Hydrogel kann funktionalisiert werden, indem man z. B. gentechnisch veränderte E. coli einbaut, die bei Induktion ein Krebsmedikament produzieren, die giftige Chemikalie Bisphenol A binden oder ihr eigenes Wachstum regulieren.
Reconstitution of contractile actomyosin rings in vesicles (Litschel et al. 2021): Die Autoren induzierten die Bildung von membrangebundenen Aktinringen in großen unilamellaren Vesikeln, die dem kontraktilen Teilungsring in vielen Zellen ähneln. Die Autoren kapselten G-Aktin mit den Aktin-Bündelungsproteinen Talin und Vinculin ein und verknüpften die Aktin-Filamente über Biotin-Neutravidin-Bindungen mit der Phospholipid-Doppelschicht. Unter Einbeziehung von Muskelmyosin bildeten sich kontraktile Aktomyosinringe, die als ein erster Schritt in Richtung eines Minimal-Divisoms für Protozellen angesehen werden können. Die Ringe können kontraktile Kräfte direkt auf die Membran leiten, was zu einer Formveränderung des Vesikels führt.
Compacting a synthetic yeast chromosome arm (Luo et al. 2021): Die Autoren reduzierten das Genom von Saccharomyces cerevisiae mit Hilfe der LoxP-sites, die im Rahmen des synthetischen Hefegenomprojekts Sc2.0 in synthetische Chromosomen integriert worden waren. Diese sites wurden integriert, um einen Prozess namens SCRaMbLE (synthetic chromosome rearrangement and modification by LoxPsym-mediated evolution) zu ermöglichen, der durch Inversion, Deletion, Duplikation und Translokation bei Expression der Cre-Rekombinase genetisch vielfältige Hefezellen erzeugt. Die Autoren integrierten selektierbare Markergene in die Sequenz zwischen zwei LoxP-sites, sogenannten LoxP-Einheiten (LU), die die Identifizierung von Stämmen ermöglichen, bei denen eine LU verloren gegangen ist. Sie testeten diese Methode mit einem Hefestamm, der den synthetischen linken Arm von Chromosom XII (synXIIL) enthält, und identifizierten einen Stamm, der 26 % (45 kbp) von synXIIL verloren hatte. Eine stärkere Reduktion des Chromosoms synXIIL war schwierig zu erreichen, da einige nicht essentielle Gene in derselben LU wie ein essentielles Gen lokalisiert waren. Um dies zu überwinden, wurde ein episomaler Gen-array mit essenziellen Genen konstruiert, um den Verlust dieser Gene auszugleichen. Auf diese Weise konnten einige LUs mit essenziellen und nicht essenziellen Genen entfernt werden, was zu einem Hefestamm führte, aus dem 58 % (100 kb) von synXIIL entfernt waren. Man geht davon aus, dass die Verkleinerung des Hefegenoms zu besseren Chassis-Organismen für die Produktion von Metaboliten führt, da weniger Energie für unnötige Prozesse verbraucht wird.
Programmable Aggregation of Artificial Cells with DNA Signals (Qiu et al. 2021): Die Autoren untersuchten die programmierbare Kommunikation und Koordination zwischen Vesikeln mithilfe von DNA-Strängen. Sie schufen große und kleine unilamellare Vesikel (giant unilamellar vesicles, GUV und small unilamellar vesicles, SUV), die auf ein DNA-Signal hin miteinander interagieren. Die GUVs besitzen Poren aus DNA-Origami, die mit planaren DNA-Origami-Kappen verschlossen sind. Die Kappen können durch einen toehold-vermittelten Strangaustausch entfernt werden, wodurch die Poren für eine externe hairpin-DNA geöffnet werden. Die hairpin-DNA kann sowohl an die GUV als auch an die SUV binden, jedoch nur, wenn sie von einem im GUV eingekapselten Enzym gespalten wird. Nach der Spaltung diffundiert die hairpin-DNA aus dem GUV heraus und bindet beide Vesikel, so dass die SUVs auf dem GUV aggregieren. Diese Aggregation kann durch einen "Freisetzungsstrang", der komplementär zur transduzierten hairpin-DNA ist, rückgängig gemacht werden. Das hier entwickelte System kann für die Zell-Zell-Kommunikation und das koordinierte Zellverhalten in der synthetischen bottom-up-Biologie verwendet werden.
2020
Biner et al. (2020) erstellten und charakterisierten Energie-regenerierende Nanovesikel (erNV), welche den Prozess der oxidativen Phosphorylierung zur ATP-Regenerierung in Mitochondrien nachahmen. Zur Erstellung der erNVs wurde eine Lipidmischung mit einem Mindestsatz an Atmungsketteneinheiten vermischt, bestehend aus Ubichinon-10 (Q10), ATP-Synthase-Komplex (Ec-F1F0) und dem Komplex I (mito-CI) aus dem Hausrind (Bos taurus) sowie der alternativen Oxidase (AOX) aus Trypanosoma brucei brucei. Dabei sollen die erNVs theoretisch in der Lage sein, die Elektronen aus der Oxidation von Nikotinamidadenindinukleotid (NADH) zu NAD+ zu nutzen, um einen elektrochemischen Protonengradienten (Δp) aufzubauen und dadurch ATP zu synthetisieren. Die experimentellen Daten aus verschiedenen Assays bestätigen, dass sich alle verwendeten Komponenten überwiegend in der funktionsfähigen Orientierung in die Vesikelmembran integrieren. Zudem sind die erNV in der Lage ATP zu produzieren. Im Vergleich mit sub-mitochondrialen oder sub-bakteriellen Vesikeln, welche aus nativen Säuger- bzw. Bakterienmembranen stammen, sind die erNV in der Lage, einen ähnlich hohen Δp aufrecht zu erhalten. Dies bedeutet konkret, dass nur 50 % der Protonen durch Membranundichtigkeiten verloren gehen und eine stabile ATP-Produktion aufrechterhalten werden kann. Diese Arbeit ist das erste Beispiel für ein semi-synthetisches System, bei dem die Oxidation von NADH zur Synthese von ATP verwendet wird. - zur Originalliteratur
Buddingh’ et al. (2020) etablierten ein interzelluläres und allosterisch aktivierbares Kommunikationsnetzwerk in artifiziellen Zellen. Um diese Kommunikationsplattform zu implementieren, wurden zwei unterschiedliche Populationen artifizieller Zellen (giant unilamellar vesicles) hergestellt. Die „Sender“-Zellen reagieren auf ein externes Signal, mit der Bildung von Adenosin-5´-Monophosphat (AMP) als Signalmolekül, welches in das umgebende Medium abgegeben wird. Die „Empfänger“-Zellen sind durch die AMP abhängige allosterische Aktivierung der Glykogenphosphorylase B (GPb) in der Lage, das Signal zu erkennen und zu verstärken. Im direkten Vergleich mit der Hintergrundaktivität bei AMP-Abwesenheit führt schon eine geringe Konzentration von AMP zu einer Konformationsänderung der GPb in den „Empfänger“-Zellen, was zu einer erhöhten Phosphorolyse von Glykogen und einem 80-fachen Anstieg der Nicotinamidadenindinukleotid (NADH)-Produktion führte. Somit wurde erstmals ein allosterisch aktivierbares Kommunikationsnetzwerk erstellt, welches vollständig von der Stoffwechselmaschinerie und kleinen Molekülen als chemische Informationsträger abhängt und zusätzlich in der Lage ist, Signale über eine größere Distanz innerhalb eines Konsortiums artifizieller Zellen zu propagieren. - zur Originalliteratur
Fan et al. (2020) generierten chromosomenfreie Zellen (SimCells) basierend auf Escherichia coli, Pseudomonas putida und Ralstonia eutropha, welche als vereinfachte synthetische Vehikel fungieren. Das Entfernen der Bakterienchromosomen erfolgte, indem mittels der heterologen Endonuklease I-Ceul Doppelstrangbrüche eingeführt wurden und anschließend ein Abbau durch endogene Nukleasen einsetzte. Die so generierten SimCell blieben weiterhin funktionsfähig und stabil. Insbesondere zeigten die SimCells eine stabile Expression von eingebrachten synthetischen Stoffwechselwegen über einen Zeitraum von zumindest zehn Tagen. In einem proof-of-principle-Experiment wurde ein Stoffwechselweg zur Synthese eines Krebsmedikamentes (Brenzcatechin) in die SimCells eingebracht, wodurch die Lebensfähigkeit von Krebszellen in Zellkultur nach Inkubation mit den SimCells signifikant verringert wurde. Somit stellen die SimCells ein synthetisches Werkzeug dar, das z. B. für medizinische Anwendungen programmiert werden kann, um Stoffe ohne unerwünschte Wechselwirkungen mit dem Wirtsgenom sicher zu synthetisieren und zu transportieren. - zur Originalliteratur
Yang et al. (2020) entwickelten Protozellen als minimalistische Kommunikationsmodelle für Zell-Zell-Kommunikationsstudien, basierend auf dem 2019 von Joesaar et al. veröffentlichten Modell. Die Autoren bauten zwei verschiedene Proteinosomen-basierte semipermeable Protozellen, die biotinylierte DNA-Komplexe enthalten, die entweder senden oder empfangen können. Bei Laserbestrahlung wird ein photospaltbarer Nitrobenzyl-linker abgespalten und gibt einen DNA-Strang frei. Dieser DNA-Strang kann die Empfängerzellen erreichen und führt dort zu einer Fluoreszenzantwort. Durch Veränderung der experimentellen Bedingungen zeigten die Autoren, dass die Signalreichweite von mehreren Faktoren wie Dichte und Permeabilität der Empfänger, extrazellulärem Signalabbau, Signalverbrauch und katalytischer Regeneration bestimmt wird. Sie erstellten auch eine Empfängerzelle mit einem UND-Empfänger, der raumzeitlich von zwei Senderzellen aktiviert wurde. - zur Originalliteratur
2019
Berhanu et al. (2019) beschreiben ebenfalls eine Form synthetischer Zellen, die sich selbst mit Energie für die Proteinsynthese versorgen können. Dazu wurden giant unilamellar vesicles mit einem künstlichen Organell konstruiert, welches die Membranproteine Bakteriorhodopsin und F-Typ ATP-Synthase enthält. Über das Bakteriorhodopsin werden Protonen in das Organell gepumpt, welche dann von der ATP-Synthase zur ATP-Produktion genutzt werden. Bei Zugabe eines modifizierten Transkriptions-Translationssystems kann das produzierte ATP als Substrat für mRNA, zur Phosphorylierung von GDP und als Energie für die Aminoacylierung der tRNA genutzt werden. Das System ist so in der Lage, Bakteriorhodopsin und Untereinheiten der ATP-Synthase de novo zu synthetisieren und somit die photosynthetische ATP-Produktion in den artifiziellen Organellen zu erhöhen. - zur Originalliteratur
Diederichs et al. (2019) - Die Autoren entwickelten Moleküle für die Bildung von Nanoporen mit einem Durchmesser von 20,5 nm und einer Höhe von durchschnittlich 31,5 nm, durch die auch gefaltete Proteine wie fluoreszierende Proteine transportiert werden können. Die Nanoporen bestehen aus quadratisch angeordneten DNA-Duplexen, die zur Insertion in die Membran mit Cholesterol-Lipidankern versehen sind. So entstehen 50 nm2 große Poren, die in artifiziellen Zellen eingesetzt werden könnten. - zur Originalliteratur
Ghosh et al. (2019) - Während des Substratumsatzes kann bei vielen Enzymen eine diffuse Bewegung beobachtet werden, die sich mit der Erhöhung der Substratkonzentration noch verstärkt. Diese Tatsache ausnutzend, produzierten Ghosh et al. Phospholipidvesikel, die als Transmembranmolekül die Na+/K+-ATPase enthielten. Anschließend wurden diese Vesikel unter physiologischen Bedingungen in Kultur getestet und zeigten eine gerichtete Bewegung, gefolgt von einer randomisierten Reorientierung. Diese Vesikel stellen einen ersten Schritt in Richtung autonomer Nanovesikel dar und können z. B. als Transportvesikel für Biomoleküle genutzt werden. - zur Originalliteratur
Zur Weiterentwicklung von synthetischen Zellen wurden mechanosensitive Mechanismen für verschiedene Anwendungen durch zwei Forschergruppen wie folgt implementiert:
Garamella et al. (2019) konstruierten Protozellen, welche auf Umwelteinflüsse reagieren und selbständig einem Reiz entgegenwirken können. Hierfür wurden synthetische Zellen mit einer Phospholipidmembran hergestellt, welche durch Inkorporation von MscL-Ionenkanälen mechanosensitiv sind. Der Intrazellularraum der synthetischen Zelle enthält sowohl Plasmide zur Expression eines bakteriellen Zytoskelettproteins (MreB) als auch ein in vitro-Transkriptions- und Translationssystem. Durch Veränderung der äußeren Umweltbedingungen, hin zu einem hypoosmolarem Exterieur, kommt es zum mechanischen Stress der Membran und somit zu einer Porenbildung durch die MscL. Daraufhin strömt ein im extrazellulären Medium befindliches Regulatormolekül ein, welches die Expression des MreB ermöglicht. Im Anschluss migriert MreB zur Membran und erhöhte dort die mechanische Robustheit. Hiermit konnte ein adaptives synthetisches Zellsystem erstellt werden, welches sich gegenüber osmolarem Stress widerstandsfähig zeigt. - zur Originalliteratur
Hindley et al. (2019) etablierten einen mechanosensitiven Signalweg, welcher auf dem sPLA2-Membran-MscL-Netzwerk von Charalambous et al. (2012) basiert. Dabei diente die kalziumabhängige lösliche Phospholipase A2 (sPLA2) als Katalysator zur Synthese von Lysophosphatidylcholin, welches in die Membran von artifiziellen Zellen eingebaut wird. Dies wiederum führt zu einer Veränderung in der Membranmechanik und zur Öffnung des Ionenkanals MscL. Darauf aufbauend erstellten Hindley et al. artifizielle Zellen, welche sowohl mechanosensitive Vesikel mit dem Fluoreszenzfarbstoff Calcein als auch inaktivierte sPLA2 enthielten. Die anschließende extrazelluläre Zugabe von alpha-Hämolysin führte zur Bildung von Poren in der äußeren Membran, nachfolgend zum Kalzium-Influx und zur Aktivierung der sPLA2. Die veränderte Mechanik der Vesikelmembran und die daraus resultierende Öffnung des MscL wurde mittels steigender Calcein-Fluoreszenz nachgewiesen. Somit konnte ein kontrollierter Kalziumeinstrom für eine zukünftige Signaltransduktion in artifiziellen Zellen etabliert werden. - zur Originalliteratur
Joesaar et al. (2019) entwickelten Konsortien synthetischer Protozellen, die über orthogonale Kanäle miteinander kommunizieren. Die Protozellen besitzen eine Membran aus Proteinosomen, die für kurze (< 100 Basen) einzelsträngige DNA (ssDNA)-Moleküle permeabel ist und die in den Zellen enthaltene DNA vor einem Abbau im Zellkulturmedium schützt. Die Zellen kommunizieren über ssDNA miteinander, indem eine Zelle eine input-ssDNA abgibt, welche in der nächsten Zelle eine andere ssDNA ersetzt, deren Sequenz einen kleinen mismatch aufweist. Diese zweite ssDNA kann dann in die nächste Zelle diffundieren. Auf diese Weise können mehrere Zellpopulationen auch bidirektional miteinander kommunizieren. - zur Originalliteratur
Venetz et al. (2019) beschreiben die Synthese eines minimalen bakteriellen Genoms des Modellorganismus Caulobacter ethensis. Dazu wurden bereits bekannte essentielle Gene des Bakteriums mithilfe eines Algorithmus neu zusammengefügt, wobei die bestehende Abfolge der Gene und deren Orientierung beibehalten wurde. Zudem wurde das Genom für die Synthese optimiert. Dazu wurden mithilfe von 10.172 Basensubstitutionen 1233 repeats, 93 homopolymere Regionen, 4342 Regionen mit hohem GC-Gehalt sowie 1045 Endonuklease-Restriktionsschnittstellen entfernt. Zudem wurden 123.141 weitere Basensubstitutionen in proteinkodierende Regionen inseriert. Das synthetische Genom (C. eth-2.0) hat eine Größe von 785 kb und enthält 676 proteinkodierende, 54 nicht-kodierende und 1015 intergenische Sequenzen. 56,1 % aller Kodons waren durch synonyme Kodons ersetzt, sodass 87,4 % aller alternativen ORFs, 95,3 % aller vorhergesagten internen Transkriptionsstartsites und 76,7 % aller ribosome stalling-Motive entfernt wurden. Da 81,5 % der Gene von C. eth-2.0 funktional waren, scheint bei diesen Genen die Primärsequenz der mRNA, die Sekundärstruktur oder der Kontext eines Kodons im Genom keinen signifikanten Einfluss auf die biologische Funktion des kodierten Proteins zu haben. - zur Originalliteratur
2018
Niederholtmeyer et al. (2018) beschreiben synthetische Protozellen mit einer porösen Membran und einem primitiven „Zellkern“. Der „Zellkern“ ist eine aus Tonmineralien hergestellte Hydrogelkammer und enthält die zelluläre DNA. Die synthetischen Zellen können über ihre poröse Membran alle Komponenten eines zellfreien Transkriptions-Translationssystem aufnehmen und die DNA ihres „Zellkerns“ exprimieren. Die gebildeten Proteine können dann in benachbarte synthetische Zellen wandern, sodass zwei Zellen miteinander über Proteine kommunizieren und ein artifizielles quorum sensing aufbauen können. - zur Originalliteratur
Shao et al. (2018) haben alle 16 Chromosomen der Bäckerhefe S. cerevisiae zu einem sog. Super-Chromosom kombiniert, in dem die nicht erforderlichen Telomere und Zentromere sowie 19 lange repetitive Sequenzen mithilfe von CRISPR-Cas9 entfernt und die übrigen Chromosomen-Abschnitte fusioniert wurden. Die Forscher um Luo et al. (2018) verfolgten dasselbe Ziel und gelangten über einen ähnlichen Ansatz zu einer Hefe, deren Genom nur noch aus zwei Chromosomen besteht. Hintergrund der Experimente war die Beobachtung, dass die Anzahl der Chromosomen in Eukaryoten eher willkürlich und unabhängig von der Menge genetischer Information zu sein scheint. Die Reduktion der Chromosomenzahl hatte in der Tat nur geringe Auswirkungen auf die meisten Eigenschaften der Hefe. Diese zeigten einen ähnlichen Phänotyp und ein nahezu identisches Transkriptom. Lediglich das Wachstum war in der Hefe mit nur einem Chromosom verlangsamt. Die Bildung von Ascosporen als Teil der sexuellen Reproduktion war für beide Stämme deutlich reduziert. Durch die Genomumorganisation wurde erreicht, dass sich der neue Organismus nicht mehr mit dem Wildtyp kreuzen kann und er somit reproduktiv isoliert ist. Hierdurch ist das klassische Kriterium für die Zuordnung zu zwei verschiedenen biologischen Spezies erfüllt. - zur Originalliteratur: Shao et al. (2018), Luo et al. (2018)
Xenobiologie
2022
Für das Jahr 2022 wurden keine Veröffentlichungen aus dem Bereich Xenobiologie zur Publikation auf der ZKBS-Homepage ausgewählt.
2021
Multiplex suppression of four quadruplet codons via tRNA directed evolution (DeBenedictis et al. 2021): Die Autoren nutzten gerichtete Evolution, um einen kleinen Satz von Quadruplett-tRNAs (qtRNAs) zu etablieren, die Aminosäuren unter Multiplex-Bedingungen einbauen. Durch die Einführung eines Quadruplett-Codons als Reporter in das β-Galactosidase-Gen wurden Codon-Anticodon-Interaktionen zunächst in E. coli validiert. Mit dieser Strategie wurden 24 qtRNAs identifiziert, die das eingeführte Codon dekodieren und infolgedessen eine bestimmte Aminosäure auf das Protein laden. Die identifizierten qtRNAs wurden durch gerichtete Evolution in Phagen weiterentwickelt. Die Phagen konnten sich nur vermehren, wenn ein bestimmtes Quadruplett-Codon entschlüsselt wurde. Die Autoren zeigten die Translation von sechs aufeinanderfolgenden UAGA-Codons sowie die Translation von vier verschiedenen Quadruplett-Codons in ein und demselben Protein. Die Arbeit ist ein weiterer Schritt auf dem Weg zu einem ausschließlichen Quadruplett-Codon-Translationssystem.
Sense codon reassignment enables viral resistance and encoded polymer synthesis (Robertson et al. 2021): Die Autoren haben sense-Codons freigemacht, um mehrere verschiedene nicht-kanonische Aminosäuren (ncAAs) einzubauen. Mit Hilfe eines veränderten tRNA-Aminoacyl-tRNA-Synthetase-Paares (aaRS) können ncAAs als Reaktion auf ein bestimmtes Codon eingebaut werden. Die Autoren verwendeten den von Fredens et al. entwickelten E. coli-Stamm Syn61, in dem zwei Serin-Codons (TCG und TCA) sowie das Stopp-Codon UAG gegen synonyme Codons ausgetauscht wurden, und entwickelten den Stamm so weiter, dass die tRNAs und der release factor 1, der TCG, TCA und TAG dekodiert, entfernt wurden. Durch die Deletion der tRNAs und des release factor wurden die Zellen resistent gegen Phagen, die diese drei Codons in ihrem Genom aufweisen. Die Codons wurden zudem für den Einbau von drei verschiedenen ncAAs in ein Protein neu zugeordnet. Der Einbau von ncAAs in Proteine kann für die Entwicklung neuer Biomaterialien oder Biotherapeutika genutzt werden.
2020
Chen et al. (2020) haben einen Escherichia coli-Stamm generiert, der autonom 5-Hydroxytryptophan (5HTP) als 21. nicht-kanonische Aminosäure (ncAA) biosynthetisiert und in Proteine einbaut. Dies stellt ein Werkzeug für die Evolution neuartiger Proteine dar, die ncAAs enthalten, und kann zur Generierung neuer therapeutischer Proteine genutzt werden. Um 5HTP als ncAA in Prokaryoten nutzen zu können, musste die Hydroxylierungsmaschinerie von Tryptophan (dem Substrat für die 5HTP-Bildung) in E. coli eingeführt werden. In einem ersten Schritt wurde dafür ein Plasmid generiert, das eine spezifische Hydroxylase (z. B. XcP4H aus Xanthomonas campestris), einen künstlichen Recyclingweg für den Hydroxylase-Kofaktor Tetrahydromonapterin (MH4) und eine Expressionskassette für das grün fluoreszierende Protein (GFP) mit einem Amber-Stoppkodon an Position Tyr151 enthält. Ein zweites Plasmid, das eine modifizierte Tryptophanyl-tRNA-Synthetase (ScTrpRS)/tRNATrp aus Saccharomyces cerevisiae enthält, stellt die bioorthogonale Maschinerie zum Einbau von 5HTP am Amber-Stoppkodon bereit. Beide Plasmide wurden in E. coli transferiert, es kam zu einer von der Hydrolase und den MH4-Recycling-Genen abhängigen Expression von GFP, wobei 5HTP effizient und spezifisch eingebaut wurde. Als proof-of-concept verwendeten Chen et al. den E. coli-Stamm zur Expression des 5HTP-haltigen singe-chain variable fragment (scFv), eines Antikörperfragments gegen den humanen epidermalen Wachstumsfaktors 2 (HER2). - zur Originalliteratur
Fischer et al. (2020) identifizierten weitere unnatürliche Kodons, die in den von Zhang et al. (2017) erstellten, semi-synthetischen Organismus (SSO) inseriert werden. Da der SSO (Escherichia coli) in der Lage ist, das unnatürliche Basenpaar (UBP) NaM und TPT3 in verschiedenen Sequenzkontexten zu replizieren und zu transkribieren, nutzten Fischer et al. dieses UBP, um eine systematische Analyse von unnatürlichen Kodons durchzuführen. Durch die Insertion des NaM/TPT3-Basenpaars können neue Kodons erzeugt werden. Um diese unnatürlichen Kodons zu testen, wurden GFP-exprimierende Plasmide erstellt, bei denen NaM oder TPT3 an erster, zweiter oder dritter Stelle des Kodons 151 inseriert wurde und welche eine tRNA mit den entsprechenden Antikodons zum Einbau einer nicht-kanonischen Aminosäure (ncAA) enthalten. Diese Plasmide wurden in E. coli eingebracht. Anhand der GFP-Fluoreszenz wurden neun Kodon-Antikodon-Paare identifiziert. Alle waren stabil in der genomischen DNA kodiert und konnten erfolgreich in eine mRNA übersetzt bzw. von ihrer spezifischen tRNA gebunden werden, wodurch eine effiziente Dekodierung am Ribosom vermittelt werden konnte. Darüber hinaus wurden drei dieser Kodon-Antikodon-Paare weiter charakterisiert, indem gleichzeitig ihre Kodons in das GFP-Gen inseriert und ihre Antikodons in tRNAs in E. coli eingebracht wurden. Die drei Kodons waren orthogonal zueinander und konnten gleichzeitig innerhalb des SSO dekodiert werden. Dadurch ist ein Organismus entstanden, der erstmals in der Lage ist, 67 Codons zu dekodieren. - zur Originalliteratur
Flamme et al. (2020) haben unnatürliche Basenpaare (UBP) enzymatisch auf der Basis von Metall-Ionen erzeugt. Diese Metall-Basenpaare sind orthogonal zu den natürlichen Watson-Crick-Basenpaaren und integrieren sich leicht in die Struktur der DNA. Der Einbau von Basenpaaren mit Metallionen erweitert die chemische Vielfalt von DNA und RNA und kann für neuartige Nanomaterialien genutzt werden. Für die enzymatische Synthese eines silbervermittelten UBP verwendeten die Autoren zwei synthetische Analoga: das imidazolhaltige Nukleosid dIMC und das 6-Pyridylpurin-Nukleosid dPurP. Dieses silbervermittelte UBP (dIMC - AgI - dPurP) wurde dann mit Hilfe eines zweistufigen Syntheseprotokolls in einen DNA-Duplex eingebaut. - zur Originalliteratur
Lee et al. (2020) untersuchten, wie der Einbau von nicht kanonischen Aminosäuren mit einem verlängerten Kohlenstoff-Rückgrat (Aminosäure-Analoga) verbessert werden kann, und machten so einen weiteren Schritt Richtung Synthese von nicht-kanonischen sequenzdefinierten Polymeren. Das Beladen der tRNA mit einem Aminosäure-Analogen ist aktuell schwierig umzusetzen und wurde mithilfe des Flexizym-System (Fx, ein Aminoacyl-tRNA-Synthetase-ähnliches Ribozym) untersucht. Es zeigte sich, dass die Beladung von Aminosäuren mit langkettigen Kohlenstoffstrukturen durch eine intramolekulare Lactambildung behindert wurde. Um dies zu umgehen, wurde die intramolekulare Lactambildung durch sterische Restriktion der Amino- und aktivierten Esterfunktionalitäten verhindert. Diese molekulare Architektur nutzt dazu einen starren spacer wie Vinyl oder eine zyklische Struktur in der zentralen Region der Aminosäure und erhöht so die Gesamtladeeffizienz des Fx-Systems. Um diese Erkenntnisse auf eine natürlichere Protein-Synthese zu übertragen, wurden die Fx-geladenen tRNAs in einem zellfreien Proteinsynthese-System eingesetzt, um die Aminosäure-Analoga während der Proteinsynthese am C- und N-Terminus einzubauen. Es konnte gezeigt werden, dass jedes Substrat, das auf tRNAs geladen werden konnte, erfolgreich durch Wildtyp- oder technisch modifizierte Ribosomen in ein Peptid eingebaut wurde. - zur Originalliteratur
Yang et al. (2020) haben die Studien von Eremeeva et al. (2017) erweitert, die die Amplifikation von DNA mit vier nicht-kanonischen Basenpaaren (DZA) zeigten. Diese chemischen Modifikationen können die Stabilität und Vielseitigkeit von Nukleinsäuren erhöhen, z. B. in der biomedizinischen Forschung. Die Autoren testeten vollständig basenmodifizierte RNA (RZA) bei der Replikation, Transkription und reversen Transkription und etablierten den Informationsfluss zwischen natürlicher und unnatürlicher genetischer Information. In vitro konnte die PCR-synthetisierte DZA in RZA oder in unmodifizierte RNA transkribiert und in DNA oder DZA revers transkribiert werden. In vivo wurde DZA in Escherichia coli transkribiert und in das rot fluoreszierende Protein mTangerine translatiert. - zur Originalliteratur
2019
Calles et al. (2019) erstellten einen genetischen fail safe code, der von der natürlichen Translationsmaschinerie gelesen werden könnte und einen protektiven Effekt gegenüber Spontanmutationen aufweist. Dabei wird jede Aminosäure nur durch ein einziges Kodon kodiert, alle anderen Kodons für diese Aminosäure wären nicht translatierbare „Null-Kodons“. Idealerweise soll dabei die Mutation eines dieser sense-Kodons nicht zu einem anderen sense-Kodon führen. Dies ist bei einem 3-Basen-Kode nur möglich, wenn lediglich 16 Aminosäuren kodiert werden. Wird allerdings ein 4-Basen-Kode verwendet, können alle 20 Aminosäuren und zusätzliche, nicht natürliche Aminosäuren kodiert werden, ohne dass eine Mutation in einem anderen sense-Kodon resultiert. In einer ersten Anwendung des fail safe code wurde ein in vitro-Transkriptions- und Translationssystem genutzt. In diesem System wurden alle natürlichen tRNAs depletiert und durch 20 tRNAs ersetzt, welche die 20 Kodons des fail safe code erkennen. Somit waren keine tRNAs vorhanden, die „Null-Kodons“ erkennen können. - zur Originalliteratur
Hoshika et al. (2019) haben den genetischen Code um vier synthetische Nukleotide erweitert und so ein Acht-Buchstaben-Alphabet generiert. Die synthetische DNA verhält sich weitestgehend wie die natürliche DNA: Sie bildet eine Doppelhelix aus, in der die Interaktion über Wasserstoffbrückenbindungen erfolgt, und kann in RNA transkribiert werden. Organismen, die dieses Alphabet nutzen und stabil weitervererben wurden noch nicht generiert. - zur Originalliteratur
Kneuttinger et al. (2019) - Die Autoren haben nicht-natürliche Aminosäuren (nnAminosäuren) in das Bienzym Imidazol-Glycerolphosphatsynthase inseriert, um die Aktivierung des Enzyms über Licht zu erreichen. Im ursprünglichen Mechanismus erfolgt die Aktivierung der HisH-Untereinheit ausschließlich durch die Bindung eines Substrates an die HisF-Untereinheit. Durch den Einbau der drei lichtempfindlichen nnAminosäuren Phenylalanin-4'-Azobenzen, o-Nitropiperonyl-O-Tyrosin und Methyl-o-Nitropiperonyllysin in die substratbindende Enzymuntereinheit wurde eine lichtinduzierbare und reversible Erhöhung der Aktivierung der HisH-Aktivität erreicht, sodass eine räumliche und zeitliche Kontrolle der Enzymaktivität durch Licht ermöglicht wird. - zur Originalliteratur
Koh et al. (2019) stellten auxotrophe Escherichia coli-Bakterien her, bei denen die β-Untereinheit des DNA-Polymerase III-Holoenzyms an Position 273 anstelle eines Leucins das synthetische p-Benzoylphenyl-Alanin (pBzF) enthält und die daher nur in Anwesenheit von pBzF im Medium wachsen können. Die auch als sliding clamp bezeichnete β-Untereinheit ist in Gram-negativen Bakterien stark konserviert, sodass der auxotrophe Stamm eine nicht detektierbare escape frequency aufweist. Die beschriebene Auxotrophie kann zudem leicht auf andere Bakterien wie Pseudomonas aeruginosa oder Acinetobacter baumanii übertragen werden, um konditional lebensfähige Lebendimpfstoffe gegen diese Krankenhauskeime herzustellen. - zur Originalliteratur
Reinkemeier et al. (2019) arbeiten an der orthologen Translation des Amber-Stopp-Kodons (TAG). Um dieses Kodon zum Einbau einer nicht-kanonischen Aminosäure in ein Protein nutzen zu können, sind eine orthogonale tRNA und eine tRNA-Synthetase nötig. Dieses tRNA/tRNA-Synthetase-Paar wurde mit einer mRNA, deren Amber-Stopp-Kodon rekodiert werden soll, in einem synthetischen Organell in räumliche Nähe gebracht. Dazu wurden das tRNA/tRNA-Synthetase-Paar sowie eine RNA-Bindedomäne mit einem sogenannten assembler-Protein fusioniert. Dies kann z. B. ein Protein sein, welches innerhalb der Zelle eine Phasentrennung durchläuft oder ein Kinesin, das sich räumlich an den Plusenden der Mikrotubuli anreichert. In dem künstlichen Organell finden sich somit sowohl das tRNA/tRNA-Synthetase-Paar als auch eine mRNA mit einer Erkennungssequenz für die RNA-Bindedomäne wieder, sodass die Neukodierung des Stopp-Kodons nur in ausgewählten mRNAs erfolgt. - zur Originalliteratur
Swenson et al. (2019) haben ein synthetisches, bilinguales Biopolymer entwickelt, welches die Eigenschaften einer Aminosäuresequenz und die einer Nukleotidsequenz in einem einzigen Peptid-Nukleinsäure (PNA)-Gerüst vereint. Der PNA-Strang besitzt dabei Aminosäure-ähnliche Seitenketten, die aufgrund ihrer Anordnung ein ähnliches Aggregations- bzw. Faltungsverhalten wie ein Peptid aufweisen. Zusätzlich trägt der PNA-Strang aber auch Nukleotide in einer bestimmten Sequenzfolge, die durch komplementäre DNA oder RNA gebunden werden können. Als Anwendung zeigten die Autoren, dass sich PNA-Stränge mit hydrophoben und hydrophilen Aminosäureseitenketten im wässrigen Milieu zu Mizellen zusammen lagern. Bei Zugabe einer zur im PNA-Strang enthaltenen Nukleinsäuresequenz komplementären RNA wurden die Mizellen wieder zerstört. PNAs sollen auch genutzt werden, um eine präzise Interaktion zwischen verschiedenen Ziel-Proteinen und -Nukleinsäuren zu vermitteln. - zur Originalliteratur
Methoden mit Einfluss auf die Synthetische Biologie
2022
Für diesen Bereich wurden sieben Publikationen ausgewählt.
Schwärmende Bakterienkolonien, die durch die Expression von Adhäsinen und ihren komplementären Antigenen miteinander interagieren und verschiedene Muster ausbilden können, wurden entwickelt. Diese können beispielsweise in diagnostischen Displays eingesetzt werden (Kim et al. 2022). Design und Konstruktion von verschiedenen metabolischen Schaltkreisen oder Bakterienstämmen können durch automatisierte Plattformen wie Galaxy-SynBioCAD erleichtert werden. Das Gemeinschaftsprojekt erlaubt es allen Interessierten, neue Werkzeuge hinzuzufügen und alte zu bewerten (Herisson et al. 2022). Auch die open-source-Plattform basicsynbio soll das Konzipieren genetischer Schaltkreise erleichtern. Es basiert auf einer standardisierten DNA-Assemblierungsmethode und ermöglicht es Nutzern, verschiedenste Schaltkreise zu entwickeln und zu exportieren (Haines et al. 2022). Um biosynthetische Gencluster für die Anwendung nutzbar zu machen, können diese in silico umgestaltet und zu neuen synthetischen genetischen Elementen zusammengefügt werden. Dadurch wird auch eine Expression in einem anderen Reich der Lebewesen möglich (Patel et al. 2022). Die Evolink-Plattform erlaubt es, zuvor konzipierte Ribosomen, deren rRNAs verbunden sind und die in der Lage sind, nicht-natürliche Aminosäuren in Proteine einzubauen, in ihrer Funktion zu verbessern. Die Plattform erstellt und testet verschiedene Nukleinsäurebibliotheken mithilfe iterativer design-build-test-Analysen (Kim et al. 2022). Das METIS (Machine-learning guided Experimental Trials for Improvement of Systems)-System wurde entwickelt, um die Lücke zwischen maschinellem Lernen und seiner Anwendung in biologischen System zu schließen. Experimentatoren brauchen keine Programmiererfahrung, um biologische Netzwerke wie den synthetischen CETCH-Zyklus zur CO2-Fixierung zu optimieren (Pandi et al. 2022). Ein maschineller Algorithmus wurde ebenso verwendet, um die Effektivität einer PET-Hydrolase zu steigern. Diese soll zum enzymatischen Abbau von Plastikabfall genutzt werden (Lu et al. 2022).
2021
Division and regrowth of phase-separated giant unilamellar vesicles (Dreher et al. 2021): Die Autoren haben einen vollständig kontrollierten Teilungsmechanismus für große unilamellare Vesikel (GUV) entwickelt. Dazu verwendeten sie die Phasentrennung, um GUVs mit einer ungeordneten und einer geordneten Lipidphase zu konstruieren. Wenn die Osmolarität des umgebenden Mediums erhöht wurde, verringerte sich das Volumen der GUVs durch Wasserabfluss und es bildeten sich zwei kleinere Vesikel. Die Technik wurde auch verwendet, um eine lichtinduzierte Vesikelteilung zu entwickeln. Zu diesem Zweck wurde dem GUV-haltigen Medium in Bis-(5-carboxymethoxy-2-nitrobenzyl)-Ether (CMNB) eingeschlossenes Fluorescein zugesetzt. Bei Beleuchtung wird diese zunächst nicht fluoreszierende Verbindung in drei Komponenten aufgespalten, so dass die Osmolarität erhöht wird und sich die GUVs teilen. Die Autoren untersuchten auch das Vesikelwachstum durch Fusion von Vesikeln mit geordneten und ungeordneten Lipiden und zeigten, dass Fusionen nur mit einer eher geringen Frequenz stattfinden. Die Fusionshäufigkeit kann durch die Verwendung von CaCl2 oder DNA-basierten zipper-ähnliche Molekülen, die das SNARE-Protein nachahmen, erhöht werden. Diese bringen die Vesikel in räumliche Nähe zueinander und treiben die Fusion voran. Zusammengenommen ermöglicht diese Methode eine kontrollierbare GUV-Teilung in zwei Kompartimente der zweiten Generation und stellt einen weiteren Schritt in Richtung synthetische Zellteilung dar.
Manufacture of multi-layered artificial cell membranes through sequential bilayer deposition on emulsion templates (Ip et al. 2021): Die Autoren entwickelten eine emulsionsbasierte Methode zur Erzeugung von großen Vesikeln mit mehreren Membranen. Zur Erzeugung dieser mehrschichtigen Vesikel wurden Wasser-in-Öl-Tröpfchen als Vorlage verwendet und nacheinander mit Lipid-Monolayern umgeben. Das innerste Monolayer-Tröpfchen wurde durch Inkubation von wässrigen Emulsionströpfchen in einer Öllösung gebildet. Weitere Monoschichten wurden hinzugefügt, indem das ursprüngliche Tröpfchen in einer Zentrifuge durch mehrere Monoschicht-stabilisierte Öl/Wasser-Grenzflächen mit abwechselnd hydrophober/hydrophiler Außenorientierung getrieben wurde. Die erzeugten Vesikel mit mehreren Schichten hatten einen Radius von 0,5 bis 50 µm und waren mechanisch stabil. Die erfolgreiche Schichtung wurde durch Zugabe verschiedener fluoreszierender Lipide (Rh-PE, NBD-PE, Cy-5-PE) zu jeder Doppelschicht kontrolliert. Zusätzlich wurde dem Vesikelmedium Natriumdithionit, ein membranundurchlässiger NBD-Quencher, zugesetzt. Aufgrund des fehlenden Quenchens konnten die Autoren zeigen, dass die inneren Membranen, die NBD-PE enthalten, durch die zusätzlich aufgetragenen äußeren Membranen geschützt sind, wodurch eine erfolgreiche und stabile Schichtung nachgewiesen werden konnte. Die Membranmechanik, wie z. B. die Biegesteifigkeit, wurde durch thermische Fluktuation der Vesikel getestet, die mittels Phasenkontrastmikroskopie nachgewiesen wurde. Das Ergebnis war, dass mit zunehmender Anzahl von Schichten die Biegesteifigkeit zunahm, was bestätigt, dass die Mehrfachschichten mechanisch gekoppelt sind. Diese Methode könnte eine potenzielle Lösung für die Einbindung einer breiten Palette von Funktionsmodulen in synthetische Vesikel darstellen.
2020
Li et al. (2020) etablierten eine skalierbare magnetische Anordnung zur Erstellung von Kolonien aus Zell-imitierenden großen unilamellaren Vesikeln (GUVs). Um eine auf Magnetfeldern basierende Koloniebildung von GUVs zu entwickeln, wurde ein Edelstahl-Netz mit einem Mikrotiterplattenmuster, welches sich in einem paramagnetischen Medium befand, mit GUVs beladen und zwischen zwei Magnete mit Nord-Süd-Ausrichtung gelegt. Die GUVs wurden magnetisch zu Kolonien zusammengefügt, die auch eine höhere Stabilität gegenüber einem osmotischen Ungleichgewicht aufwiesen als einzelne GUVs. Um die strukturelle Komplexität von biologischem Gewebe mit unterschiedlichen Zelltypen nachzuahmen, wurden zwei Arten von GUVs mit unterschiedlichen Zusammensetzungen erzeugt und magnetisch manipuliert. In Abhängigkeit von der Richtung des Magnetfeldes, konnten die verschiedenen GUV-Arten voneinander getrennt und dabei entweder in unterschiedlichen Richtungen oder in unterschiedlichen Schichten sortiert werden oder sie bildeten eine serielle Verteilung innerhalb der Mikrowells. In einem proof-of-principle-Experiment wurden zwei unterschiedliche GUV-Arten generiert und magnetisch voneinander getrennt. Die GUVs in der Kolonie 1 enthielten Poren und wurden in Gegenwart von Glukose-Oxidase gebildet, während die GUVs der Kolonie 2 zusammen mit der Meerrettichperoxidase gebildet wurden. Zum Medium hinzugegebene Glukose gelangte über die Poren in die GUVs der Kolonie 1, H2O2 wurde produziert und diffundierte in die GUV der Kolonie 2, in denen die Meerrettichperoxidase die Produktion eines roten Fluorophors katalysierte. Da das rote Fluorophor ausschließlich in GUVs der Kolonie 2 nachgewiesen werden konnte, wurde gezeigt, dass das System in der Lage ist, die Kompartimentierung und die räumliche Trennung biochemischer Reaktionen natürlichen Gewebes rudimentär nachzuahmen. Diese Arbeit könnte die Erstellung und Untersuchung Gewebe-ähnlicher Strukturen ermöglichen und stellt einen wichtigen Schritt hinsichtlich synthetischer Gewebe dar. - zur Originalliteratur
2019
Hamashima et al. (2019) erweiterten, aufbauend auf den Arbeiten von Kimoto et al. (2013), eine Sequenziermethode zur präzisen Lokalisation der unnatürlichen Base Ds: (7-(2-Thienyl)-imidazo-[4,5-b]pyridin) in Aptamer-Kandidaten aus ExSELEX-Bibliotheken (genetic alphabet expansion for systematic evolution of ligands by exponential enrichment). Hierfür wurden zwei unterschiedliche replacement-PCRs verwendet, um unnatürliche Basen gegen natürliche Basen (N) auszutauschen, mit dem Ziel, die so generierten Produkte mittels deep sequencing zu untersuchen. Um Rückschlüsse auf die tatsächliche Position von Ds ziehen zu können, wurde eine Enzyklopädie erstellt, welche die Häufigkeit und Zusammensetzung der Konversion bei einem bestimmten Sequenzkontext (NNNDsNNN) beinhaltet. Durch Abgleich der aktuellen Daten mit den Enzyklopädiedaten konnte somit auf die Position von Ds rückgeschlossen werden. - zur Originalliteratur
Koch et al. (2019) fusionierten DNA-Moleküle mit funktionellen Materialien, um einen stabilen Datenspeicher ohne Informationsverlust zu generieren (DNA-of-things storage architecture). Hierfür wurden die DNA-Moleküle in eine Kapsel aus Silica-Nanopartikeln eingebettet, um einen Abbau der DNA im Herstellungsprozess des Speichers zu verhindern. In einem proof of priciple-Experiment wurde die in Silica enkapsidierte DNA (SPED) in Polycaprolacton (PCL), einem biologisch abbaubaren Thermoplastik-Polyester, eingebettet und mittels 3D-Druck in die Form eines Hasen gedruckt. Anschließend wurde an einer randomisierten Stelle des Hasen ein Teil entnommen und die darin befindliche DNA extrahiert, amplifiziert und dekodiert. Die im Teil des Hasen gespeicherte Datei konnte exakt wiederhergestellt werden und diente als Vorlage zum Druck der F1-Generation. Auch in den nachfolgenden Generationen bis F5 wurden keine Datenverluste beobachtet. Nachfolgende Experimente, bei denen die SPED Videodaten enthielten und in verschiedenste Materialen eingebettet wurden, bestätigten die Stabilität der SPED. Somit ist ein System entstanden, welches z. B. für die Aufbewahrung sensibler Daten über Menschengenerationen hinweg genutzt werden könnte. - zur Originalliteratur
Lee et al. (2019) - Die Autoren erweiterten das Wissen zu Flexizymen, die genutzt werden, um neuartige chemische Monomere an tRNAs zu binden. Die tRNAs bringen die Monomere zu den Ribosomen, sodass diese als nicht natürliche Aminosäuren in Peptide eingebaut werden können. Lee et al. erstellten erstmals definierte Design-Regeln für die neuartigen Monomere, damit diese ohne aufwändigen trial-and-error-Prozess einfach an tRNAs gebunden werden können. So kann eine Vielfalt an Monomeren durch Ribosomen zu Peptid-Hybridprodukten zusammengefügt werden. Als Anwendung dieser Umprogrammierung des genetischen Codes ist die Synthese neuartiger (biologischer) Materialien, z. B. high-performance Materialen, oder neuartiger Medikamente anvisiert. - zur Originalliteratur
Zhang et al. (2019) entwickelten eine Methode zur Sicherung der Daten in DNA-Datenspeichern, welche sich vom „Auslesen“ der DNA mittels Sequenzierung abkoppelt. Die in dieser Arbeit verwendete Origami-Kryptographie basiert auf dem self assembly von Biomolekülen. Für die Verschlüsselung des DNA-Datenspeichers durch den Versender wird ein langer scaffold-DNA-Strang mit message-Strängen beladen, welche ein Biomolekül wie z. B. Biotin enthalten und sich je nach zu kodierender Information in der Anzahl und Position am scaffold unterscheiden. Die Entschlüsselung der Nachricht beim Empfänger erfolgt mittels hunderter, nachrichtspezifischer scaffold-Stränge, welche den scaffold in eine bestimmte DNA-basierte Nanostuktur falten. Diese Nanostruktur ähnelt einem QR-Code mit quadratischer Form, aber mit einem Brailleschrift-ähnlichen Muster, bestehend aus mehreren Punkten. Die Orientierung des Quadrates, welche zum Auslesen der Daten wichtig ist, wird durch einen marker-DNA-Strang gewährleistet. Dieser wird vom Versender während der Verschlüsselung zu den message-Strängen hinzugegeben und befindet sich an einer dezidierten Position am scaffold. Das Auslesen der Daten erfolgt anschließend mikroskopisch unter Zugabe von Streptavidin, wobei ein Braille-Punkt einer binären Zahl entspricht und bei Textnachrichten entweder einen Buchstaben oder dessen Position repräsentiert. Somit ist erstmals ein auf Biomolekülen basierter und kryptographisch flexibler QR-Code-ähnlicher DNA-Datenspeicher entstanden. - zur Originalliteratur
Zhang et al. (2019) - Im Bereich der synthetischen Biologie ist in den letzten Jahren eine erhebliche Menge genetischer Daten entstanden, welche wiederum zur Konstruktion von genetischen Schaltkreisen eingesetzt werden können. Diese Daten sind über unterschiedliche Plattformen für die Anwender zugänglich. Sequenzbasierte Daten, bestehend aus bekannten und natürlichen Sequenzen, können über Datenbanken mit dem Basic Local Alignment Search Tool (BLAST) abgefragt werden. Komplexere synthetische Schaltkreise und genetische Designs werden in Repositorien wie SynBioHub vorgehalten und öffentlich geteilt. Hierfür verwendet SynBioHub große Datensätzen verschiedener Plattformen, wie z. B. International Genetically Engineered Machine (iGEM). Zum standardisierten Datenaustausch sind die Daten in der Synthetic Biology Open Language (SBOL) kodiert. Da sich die Datensätze der verschiedenen Plattformen, welche SynBioHub speisen, qualitativ unterscheiden und zum Teil unstrukturiert in SBOL übersetzt werden, sind Suchanfragen und genetische Designs oft unpräzise. Aus diesem Grund implementierten Zhang et al. Organisationstechniken zur Sortierung von Informationen aus dem world wide web in SynBioHub. Ziel der Arbeit war es, die Suchergebnisse bei Suchanfragen zu brauchbaren Schaltkreisen und deren Einzelkomponenten zu präzisieren. Hierfür wurden Methoden miteinander kombiniert, welche die Rangfolge der Popularität von Schaltkreiselementen festlegen (PageRank) und die Gewichtung auftretender Duplikate verringern (clustering with data infrastructure). Das in dieser Arbeit entwickelte Programm SBOLExplorer ist als backend service in SynBioHub implementiert. Der Anwender kann daher wie gewohnt den Schaltkreis graphisch über die Applikation SBOL-Designer in SynBioHub zusammenstellen und erhält bei einer Suchanfrage automatisch die sortierten Informationen. - zur Originalliteratur
2018
Bourgeois et al. (2018) haben ein CRISPR-Cas9-Integrationssystem entwickelt, das eine Reihe synthetischer DNA landing pads nutzt, um mehrere Kopien einer DNA in das Genom von S. cerevisiae zu integrieren. Die pads bestehen aus spezifischen gRNA-Sequenzen mit flankierenden rekombinogenen Regionen und fungieren als Zone für den Multi-Kopien-Gentransfer. Dabei können unterschiedliche pads einmal, zweimal, dreimal oder viermal im Hefegenom vorliegen. Dadurch ist es möglich, in nur einer Reaktion bis zu vier Kopien eines Gens präzise in das Genom der Hefe zu integrieren. In einem proof of principle-Experiment wurde ein Enzym (Norcoclaurin-Synthase) mit geringer katalytischer Aktivität gewählt, welches zur Synthese von Benzylisoquinolinalkaloiden genutzt wird. Durch Integration von vier Kopien des Enzymgens in die landing pads konnte die Produktion des Intermediatmoleküls ((S)-Norcoclaurin) zur Synthese des Alkaloids verdoppelt werden. - zur Originalliteratur
Synthetische Biologie mit Anwendungsperspektiven
2022
Für diesen Bereich wurden zwei Publikationen ausgewählt.
Ein synthetisches Transkriptionssystem steuert die Expressionsstärke eines gene of interest über verschiedene gRNAs, ihre komplementären Bindestellen sowie ein deaktiviertes CRISPR-Protein, welches an eine Aktivierungsdomäne fusioniert wurde. Dadurch ist eine präzise Expression über einen Bereich von mehr als 1000 Einheiten möglich (Chen et al. 2022). Ein oszillierender Schaltkreis, der über zwei inducer in E. coli gesteuert wird, kann sowohl seine Periode als auch seine Amplitude verändern und kann beispielsweise für eine pulsierende Verabreichung von Medikamenten verwendet werden (Zhang et al. 2022).
